Eine eigene Proteinstruktur zum 50. Geburtstag von Andrei N. Lupas
Wissenschaftler vom Max-Planck-Institut für Entwicklungsbiologie in Tübingen entwarfen eine Proteinstruktur mit einer Aminosäuresequenz, die den Buchstaben „Andrei N. Lupas“ entspricht. Der Direktor der Abteilung Proteinevolution erhielt damit die erste personalisierte Proteinstruktur zu seinem 50. Geburtstag im vergangenen Jahr. Vorgestellt wurde die wissenschaftliche Arbeit bereits im vergangenen Oktober auf dem 6. Alpbach Coiled-Coil-Workshop. Das Journal of Structural Biology veröffentlichte nun den entsprechenden Artikel auf seiner Webseite.
Etwa vier Monate dauerte es, bis die Wissenschaftler um Marcus Hartmann und Birte Hernandez Alvarez vom Max-Planck-Institut für Entwicklungsbiologie, Abteilung Proteinevolution, die passende Aminosäuresequenz in ein Protein eingebaut hatten und experimentell bestätigen konnten, dass dieses Protein die geplante Struktur aufweist. Gerade rechtzeitig war damit die Überraschung perfekt: Der Direktor ihrer Abteilung, Andrei N. Lupas, erhielt zu seinem 50. Geburtstag seine eigene und die bislang einzige personalisierte Proteinstruktur.
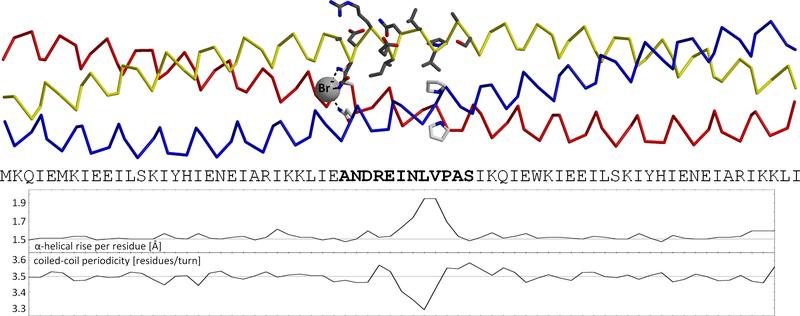
Die Wissenschaftler nutzten dafür die Stabilität eines Teils des Hefe-Transkriptionsfaktors GCN4, einer sogenannten Coiled Coil. Das sind grundlegende fiberartige Proteinstrukturelemente, die aussehen wie zu Seilen verdrillte Fasern und die in unzähligen Proteinen aller Lebensformen vorkommen. Sie bestehen aus mindestens 2 umeinander gewundenen α-Helices, welche in ihrer Aminosäuresequenz im einfachsten Fall einem periodischen Muster mit sieben Positionen folgen, dem sogenannten Heptadenrepeat. In diesen sieben Positionen ergeben sich aus strukturellen Gründen unterschiedliche Präferenzen für bestimmte Arten von Aminosäuren.
Bei der hier beschriebenen Proteinstruktur haben die Wissenschaftler die Aminosäuresequenz „ANDREINLVPAS“ zwischen zwei solche stabilisierende GCN4-Adaptoren eingefügt. „Da das ganze Konstrukt dem einfachen Heptadenrepeat folgen sollte, mussten wir dafür Sorge tragen die Sequenz so einzubauen, dass die einzelnen Aminosäuren zu den Positionen in dem zugrunde gelegten periodischen Muster passen,“ erklärt Marcus Hartmann. Er und seine Kollegen wählten als Grundlage eine Coiled Coil mit drei Helices, da dies am besten mit dem N aus Andrei zu vereinbaren war. Das V wurde anstelle des U gewählt, weil das U nicht im Aminosäurealphabet vorkommt.
Schwierig wurde es beim Buchstaben P, da die entsprechende Aminosäure Prolin eigentlich nicht in α-Helices hineinpasst. „Hier haben wir nur hoffen können, dass eine vernünftig gefaltete Struktur dabei herauskommt,“ so Hartmann. „Am Ende haben wir viel über die Unterbringung von Prolin in Coiled Coils gelernt.“ Die tatsächliche Struktur wurde mittels Röntgenkristallographie bestimmt.
Andrei Lupas hat wesentlich zum heutigen Kenntnisstand über Coiled Coils beigetragen und schuf somit auch unbeabsichtigt einen Teil der theoretischen Grundlagen zu seiner eigenen Geburtstagsüberraschung – eine eigens für ihn am Reißbrett entworfene Coiled Coil. Trotz einiger Unwägbarkeiten wie bei der Einbindung des Prolins haben es die Wissenschaftler im ersten Anlauf geschafft. Die Proteinstruktur ist nun in der Protein-Datenbank (PDB) zu finden.
Originalpublikation:
Deiss, S., et al. Your personalized protein structure: Andrei N. Lupas fused to GCN4 adaptors. J. Struct. Biol. (2014), http://dx.doi.org/10.1016/j.jsb.2014.01.013
Weitere Informationen:
http://dx.doi.org/10.1016/j.jsb.2014.01.013
http://www.eb.tuebingen.mpg.de/de/forschung/abteilungen/proteinevolution.html
Die semantisch ähnlichsten Pressemitteilungen im idw