Zellfreie Suche nach neuen Antibiotika durch Kopplung von Synthetischer Biologie mit KI
Die zunehmende Resistenz von Bakterien gegen Antibiotika stellt eine wachsende globale Gesundheitsbedrohung dar. Forschende des Max-Planck-Instituts für terrestrische Mikrobiologie in Marburg entwickelten nun mit Hilfe der Synthetischen Biologie und Künstlicher Intelligenz (KI) einen effizienteren Ansatz zur Entdeckung und Herstellung neuer antimikrobieller Peptide, die gegen ein breites Spektrum von Bakterien wirken können.
Bioaktive Peptide spielen eine Schlüsselrolle in Gesundheit und Medizin. Derzeit sind mehr als 80 peptidbasierte Medikamente im Einsatz, die alle aus natürlichen Quellen isoliert wurden. Es wird jedoch geschätzt, dass Antibiotikaresistenzen jedes Jahr weltweit mehr als eine Million Todesfälle verursachen. Es wird erwartet, dass diese Zahl bis 2050 auf 10 Millionen ansteigen wird, so dass dringend neue Methoden benötigt werden, um die Entwicklung neuer antimikrobieller Wirkstoffe zu beschleunigen. Ein ungenutztes Potenzial liegt im nicht-natürlichen Bereich, wo schätzungsweise eine Anzahl von 20 hoch 10 bis zu 20 hoch 30 verschiedene Peptide noch unerschlossen sind.
In Zusammenarbeit mit mehreren Labors am MPI für terrestrische Mikrobiologie, der Universität Marburg, dem MPI für Biophysik, dem Institut für Mikrobiologie der Bundeswehr, dem iLung Institut und INRAe Frankreich hat ein Team von Wissenschaftlern des Max-Planck-Instituts unter der Leitung von Prof. Tobias Erb eine neue Pipeline für die Entwicklung bioaktiver Peptide entwickelt.
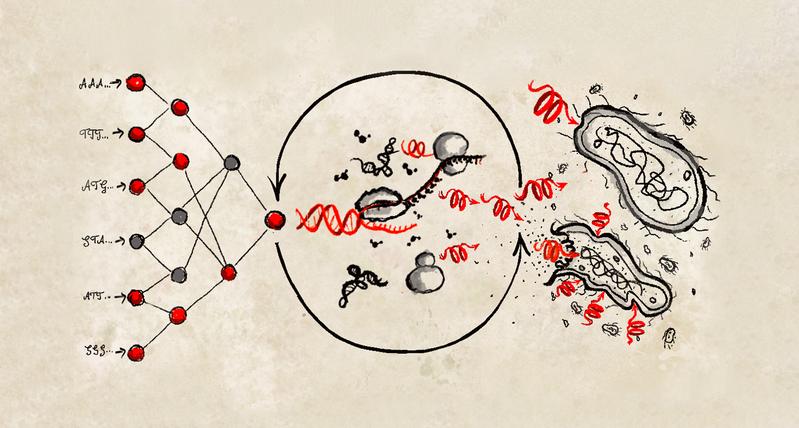
"Beim Deep Learning lernt ein neuronales Netzwerk mit Algorithmen, die durch das menschliche Gehirn inspiriert sind, aus großen Datenmengen. Diese Art des maschinellen Lernens ist für die Entdeckung von Peptiden und für das De-novo-Design sehr vielversprechend. In der Regel folgt darauf jedoch die chemische Herstellung von Peptiden für experimentelle Tests, was recht schwierig und zeitaufwändig ist. Die Zahl der Peptide, die chemisch hergestellt werden können, ist daher sehr begrenzt", erklärt Amir Pandi, Doctoral Student in der Arbeitsgruppe von Prof. Dr. Tobias Erb und Erstautor der Studie.
Um diese Einschränkungen zu überwinden, entwickelte das Forscherteam eine zellfreie Proteinsynthese (CFPS) für die schnelle und kostengünstige Herstellung von antimikrobiellen Peptiden (AMPs) direkt aus DNA-Templates. Das neue Protokoll bietet eine effiziente und kostengünstige Hochdurchsatzmethode für das AMP-Screening.
Das Team verwendete zunächst sogenanntes generatives Deep Learning, um Tausende von AMPs de novo zu entwerfen, und anschließend prädiktives Deep Learning, um diese auf 500 Kandidaten einzugrenzen. Von diesen Kandidaten wurden durch das Screening mit der zellfreien Pipeline 30 funktionelle AMPs identifiziert, die die Forscher durch Molekulardynamiksimulationen und die Bestimmung ihrer antimikrobiellen Aktivität und Toxizität weiter charakterisierten.
Bemerkenswerterweise zeigten sechs der AMPs ein breites Wirkungsspektrum gegen multiresistente Erreger und es kam zu keiner bakteriellen Resistenzentwicklung.
"Wir haben von der Kombination aus zellfreier synthetischer Biologie, künstlicher Intelligenz und Hochdurchsatzverfahren sehr profitiert. Indem wir die Anzahl der Kandidaten erhöhen, die in weniger als 24 Stunden experimentell getestet werden können, steigt die Chance, aktive AMPs zu finden", sagt Amir Pandi. "Unsere CFPS-Pipeline ergänzt nicht nur die jüngsten Fortschritte im computergestützten Wirkstoffdesign. Sie hat auch das Potenzial, die Beziehung zwischen Design und Funktion bioaktiver Peptide schneller und kostengünstiger zu erforschen". Tobias Erb fügt hinzu: "Diese neue Methode an der Schnittstelle von synthetischer Biologie und maschinellem Lernen wird für Wissenschaftlerinnen und Wissenschaftler interessant sein, die in den Bereichen Biomedizin und bioaktiven Peptidengineering arbeiten."
Zu den nächsten Schritten gehören die weitere Verbesserung der Ausbeute bei der Peptidproduktion sowie der Einsatz von KI und Ansätzen der synthetischen Biologie, um neue AMPs zu entwickeln, die stabiler und weniger toxisch sind oder eine spezifische Wirkungsweise haben. Die Forscher planen auch den Einsatz erweiterter generativer KI-Modelle. Dabei lernt die Maschine molekulare Repräsentationen für gewünschte Eigenschaften, was die Erfolgsquote bei der Identifizierung von Wirkstoffkandidaten erhöhen würde.
Wissenschaftlicher Ansprechpartner:
Prof. Dr.Tobias Erb
Prof. Dr. Tobias Erb
Director +49 6421 178-700
toerb@mpi-marburg.mpg.de
Max-Planck-Institut für terrestrische Mikrobiologie, Marburg
Originalpublikation:
Pandi; A.; Adam, D.; Zare, A.; Trinh, V.T.; Schaefer, S.L.; Burt, M.; Klabunde, B.; Bobkova, E.; Kushwaha, M.; Foroughijabbari, Y.; Braun, P.; Spahn, C.; Preußer, C.; Pogge von Strandmann, E.; Bode, H.B.; v. Buttlar, H.; Bertrams, W.; Jung, A. L.; Abendroth, F.; Schmeck, B.; Hummer, G.; Vázquez, O.; Erb. T.
Cell-free biosynthesis combined with deep learning accelerates de novo-development of antimicrobial peptides.
Nature Communications 14, 7197 (2023)
DOI https://doi.org/10.1038/s41467-023-42434-9
Ähnliche Pressemitteilungen im idw