Eine neue mathematische Sprache für biologische Netzwerke
Neues mathematisches Modell zu genetischer Interaktion identifiziert Hauptregler in biologischen Netzwerken
Ein Forscherteam um den Berliner Mathematikprofessor Michael Joswig stellt ein neuartiges Konzept zur mathematischen Modellierung der genetischen Interaktion in biologischen Systemen vor. Gemeinsam mit Biolog*innen von der ETH Zürich und Carnegy Science (USA) wurden Hauptregulatoren im Kontext eines gesamten genetischen Netzwerkes identifiziert. Die Forschungsergebnisse liefern einen kohärenten theoretischen Rahmen zur Analyse biologischer Netzwerke und wurden in den „Proceedings of the National Academy of Sciences” (PNAS) veröffentlicht.
Gemeinsame Pressemeldung mit dem Berliner Exzellenzcluster MATH+
Biolog*innen versuchen seit langem, die Schlüsselgene und -spezies zu identifizieren, die sich entscheidend auf Evolution, Ökologie und Gesundheit auswirken. Durch die Analyse entsprechender Netzwerke gelang es, bestimmte Gene als Hauptregulatoren zu identifizieren. Diese üben innerhalb des Systems eine stärkere Kontrolle aus und steuern wesentliche zelluläre Prozesse. Bisherige Studien konzentrierten sich hauptsächlich auf paarweise Wechselwirkungen innerhalb des Systems, die jedoch je nach genetischem Hintergrund oder biologischem Kontext stark beeinflusst werden können. „Kontextabhängige Effekte sind in der Biologie weit verbreitet, aber nicht ausreichend untersucht. Eine große Herausforderung bei biologischen Netzwerken ist, dass diese hochdimensional sind. Deshalb verfolgen wir mit unserem Team erstmals einen weitreichenderen Ansatz, der Wechselwirkungen höherer Ordnung einbezieht und somit Hauptregulatoren im Kontext des gesamten Netzwerkes identifiziert“, so Michael Joswig, der Professor für Diskrete Mathematik und Geometrie an der Technischen Universität Berlin, Distinguished Fellow des Berliner Exzellenzcluster MATH+ und Gruppenleiter am Max-Planck-Institut für Mathematik in den Naturwissenschaften in Leipzig ist.
Konzept der Epistase als Ansatz für hochdimensionale geometrische Modellierung
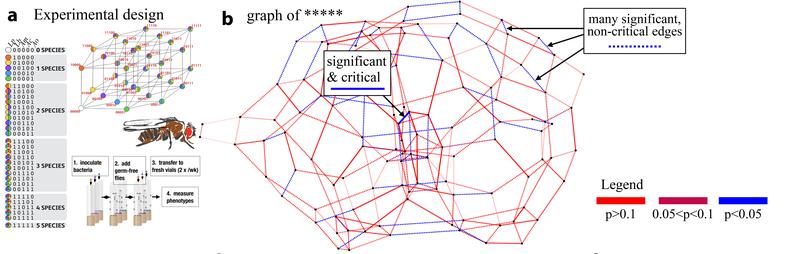
Reale Datensätze lieferten den Wissenschaftler*innen Experimente von Biolog*innen, die die Lebenserwartung der Fruchtfliege Drosophila anhand des Vorhandenseins bestimmter Bakterienkombinationen im Darm untersuchten. Um diese mathematisch zu beschreiben, nutzten die Forscher*innen einen hochdimensionalen Ansatz aus der Geometrie, der das in der Biologie bekannte Konzept der Epistase neu interpretiert. Epistase bezieht sich auf ein Wechselwirkungsphänomen zwischen verschiedenen Genen, bei dem ein Gen das Erscheinungsbild eines anderen beeinflussen kann. Diese Wechselwirkungen sind wichtig, um die genetische Vererbung und die Vielfalt der Phänotypen zu verstehen, da sie zeigen, wie verschiedene Gene miteinander interagieren, um die Expression von Merkmalen zu regulieren. Epistatische Wechselwirkungen sind in der Natur von großer Bedeutung, unter anderem hat die Epistase zwischen Bakterien im Mikrobiom weitreichende funktionelle Konsequenzen.
Die Forscher*innen betrachteten das Mikrobiom eines kompletten Stammes von Fruchtfliegen mit fünf verschiedenen Bakterienarten zuzüglich der Messungen hinsichtlich der Lebenserwartung der Fliegen bei bestimmten Kombinationen dieser Bakterien, was den Phänotyp repräsentiert. Relevante biologische Informationen wurden mittels adaptiver Landschaften, sogenannter Fitness Landscapes, kartiert und deren Epistase quantifiziert, um zu untersuchen, wie einzelne Gene und Arten die Wechselwirkungen im gesamten biologischen Netzwerk beeinflussen. Die darauf basierenden 5-dimensionalen Datensätzen wurden erneut ausgewertet und grafisch dargestellt.
Identifikation relevanter Signale als Hauptregulatoren des Netzwerkes
Der wesentliche Beitrag dieser Arbeit, die die bisherigen Ergebnisse der Forscher*innen in einer neuen Sprache beschreibt, aber auch die Vorarbeiten dazu von Niko Beerenwinkel, Lior Prachter und Bernd Sturmfels neu interpretiert, ist die Darstellung der Fitness Landscapes als epistatische Filtrierung des Netzwerkes. Diese ermöglicht es, konkrete Experimente zu analysieren, um relevante biologische Informationen zu kodieren, lesbar zu machen und in höheren Dimensionen relevante Signale zu identifizieren, die als Hauptregulatoren des Netzwerkes fungieren.
Die Studie an der Schnittstelle von Biologie und Mathematik bearbeitet eine ganze Reihe realer Experimente, um zu belegen, dass die vorgeschlagene Methode imstande ist, biologisch relevante Informationen und ihre sicheren Signale zu detektieren, aber falsch-positive Signale zu vermeiden. Dies bildet einen kohärenten theoretischen Rahmen zur Analyse biologischer Netzwerke.
Kohärenter theoretischer Rahmen zur Analyse ganzer Netzwerke
Im zuvor beschriebenen Fruchtfliegen-Experiment ist die gesamte genetische Interaktion in fünf verschiedenen Gentypen bzw. Bakterien enthalten. Jeder Genotyp weist eine Kombination von 32 Genen auf und die angewandte Methode lässt Rückschlüsse auf die Lebenserwartung der Fliege zu. Es stellt sich die Frage, was passiert, wenn zusätzliche Gene oder Parameter auftreten, was ein häufiges reales Szenario ist. Wegen ihres kohärenten geometrischen Überbaus bietet die neue Methode einen flexiblen Rahmen, auch abweichende Szenario zu detektieren und zu beschreiben. Dies war in früheren Experimenten und anderen mathematischen Modellierungen bislang nicht möglich und erforderte eine jeweils gesonderte Betrachtung der einzelnen Experimente.
„Wir freuen uns, dazu beizutragen, biologische Ergebnisse in einer mathematischen Sprache zu beschreiben. Und wir hoffen, mit unserer Methode der geometrisch-statistischen Analyse ein starkes Instrument zur Erforschung biologischer Netzwerke in höheren Dimensionen an die Hand zu geben. Es hat sich als eine hervorragende Möglichkeit zur Identifikation der Hauptregulatoren von Netzwerken erwiesen. Mit der Anwendung des neuen hochdimensionalen geometrischen Ansatzes, der die Epistase in einer Fitness Landscape quantifiziert, konnten wir entschlüsseln, wie einzelne Gene und Arten die Interaktionen im restlichen biologischen Netzwerk beeinflussen“, fasst Michael Joswig abschließend zusammen.
Die mikrobiotische Zusammensetzung im Darm hat erheblichen Einfluss auf die Lebenserwartung und es wäre zu wünschen, diese neue Quantifizierungsmethode auch auf den Menschen anzuwenden. Aufgrund der hohen Bakterienanzahl im menschlichen Darm ist dies derzeit jedoch noch nicht möglich. Wenn dies einmal mit einfacheren Methoden in Kombination mit klassischen Transformationsprozessen gelingen sollte, wäre dies der Ausgangspunkt bspw. für die Entwicklung individueller Medikamente.
Wissenschaftlicher Ansprechpartner:
Prof. Dr. Michael Joswig joswig@math.tu-berlin.de
https://page.math.tu-berlin.de/~joswig/
Originalpublikation:
Holger Eble, Michael Joswig, Lisa Lamberti und William B. Ludington
Master regulators of biological systems in higher dimensions
In Proceedings of the National Academy of Sciences” (PNAS)
DOI: https://doi.org/10.1073/pnas.2300634120
Ähnliche Pressemitteilungen im idw