Es liegt in unseren Genen – und wie sich unser Genom in 3D faltet
Ob wir gesund bleiben oder schwer erkranken, wird von unseren Genen mitbestimmt. Aber auch die Faltung unseres Genoms hat darauf maßgeblich Einfluss, denn die 3D-Genomorganisation regelt, welche Gene an- und abgeschaltet werden. Forschenden um Marieke Oudelaar und Elisa Oberbeckmann am Max-Planck-Institut (MPI) für Multidisziplinäre Naturwissenschaften ist es jetzt gelungen, die 3D-Faltung des Hefegenoms im Labor nachzustellen und die zugrunde liegenden Mechanismen zu entschlüsseln.
Unser Körper besteht aus hunderten verschiedenen Zelltypen, die auf unterschiedliche Aufgaben spezialisiert sind: Sie bekämpfen Krankheitserreger, transportieren Sauerstoff, produzieren Insulin oder lassen uns denken. Zwar enthalten alle Zelltypen den gleichen Bauplan, der in den Genen in unserer DNA verschlüsselt ist. Aber es werden nur diejenigen Gene aktiviert, die die jeweilige Zelle für ihre Aufgabe benötigt.
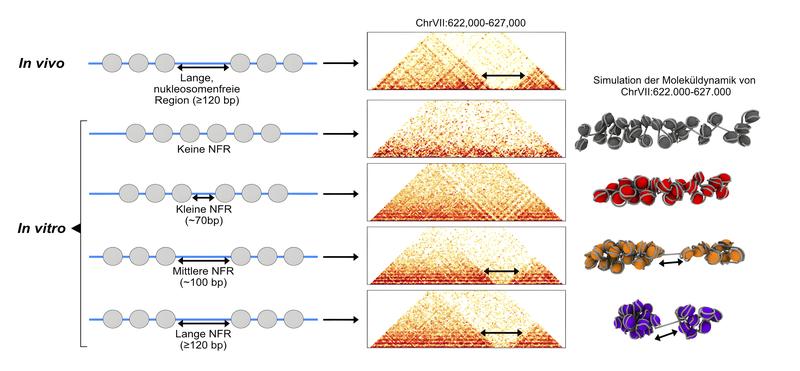
Die DNA einer Zelle unseres Körpers, unser Genom, wäre ausgestreckt etwa zwei Meter lang. Dennoch passt die DNA in einen winzigen Zellkern von rund einem Tausendstel Millimeter Durchmesser. Dafür wird sie kompakt verpackt. Bestimmte Proteine, die Histone, wickeln die DNA abschnittsweise wie auf einer Kabeltrommel auf. Dieser Histon-DNA-Komplex wird als Nukleosom bezeichnet. Die Nukleosomen sind durch nukleosomenfreie DNA-Abschnitte voneinander getrennt, ähnlich wie Knoten in einem Faden aneinandergereiht sind. Das zu Nukleosomen verpackte Genom nennt man Chromatin.
Zugängliche und aktivierbare Gene
„Die 3D-Struktur des Genoms beeinflusst die Aktivität der Gene. Durch die Faltung kommen regulatorische DNA-Abschnitte zur richtigen Zeit mit den richtigen Genen in Kontakt und schalten diese an oder aus“, erklärt Oudelaar, die am Institut eine Lise-Meitner-Forschungsgruppe leitet. Ist die Genaktivierung gestört, kann dies verschiedene Krankheiten beim Menschen wie beispielsweise Krebs verursachen.
Für Wissenschaftler*innen ist es noch immer eine Herausforderung, die 3D-Struktur der DNA zu erforschen: „In lebenden Zellen ist es schwierig aufzudecken, welche Proteine und Prozesse an der 3D-Genomfaltung mitwirken. Die zugrunde liegenden biochemischen Netzwerke sind schwer zu entwirren und die beteiligten Proteine haben häufig mehrere, schwer zu trennende Funktionen“, erklärt Elisa Oberbeckmann, Erstautorin der Studie und Projektgruppenleiterin am MPI.
Chromatin im Labor nachgebildet
Die Forscherinnen erzielten jetzt mit ihrem Team einen entscheidenden methodischen Fortschritt: Ihnen gelang es, Chromatin aus der Hefe im Labor nachzubilden und deren 3D-Struktur zu messen. „Dies bietet den großen Vorteil, dass sich der Faltungsprozess und die beteiligten Proteine isoliert untersuchen lassen“, so Oberbeckmann. Mithilfe biochemischer und genetischer Experimente sowie Computersimulationen konnten sie dann entschlüsseln, wie sich das Chromatin in bestimmte 3D-Strukturen, Chromatin-Domänen genannt, faltet.
Gaben die Forschenden im Laborexperiment Regulatoren hinzu, die Chromatin remodellieren, bildete das künstlich erzeugte Chromatin ganz ähnliche 3D-Strukturen aus wie das Chromatin in der Hefe. „Zu unserer Überraschung faltete sich dieses selbstständig, sobald zelluläre Regulatoren eine gewisse Regularität in das Chromatin gebracht haben. Die regelmäßige Anordnung der Nukleosomen durch Chromatin-Remodellierung scheint eine weit zentralere Rolle bei der Faltung des Genoms zu spielen als bisher gedacht und bestimmt maßgeblich dessen 3D-Organisation“, erklärt Oudelaar.
Wie das Team auch zeigen konnte, bilden lange nukleosomenfreie Abschnitte im Chromatin dabei die Grenzen zwischen den einzelnen Domänen. „Die Länge dieser Abschnitte hat direkten Einfluss, ob benachbarte Domänen miteinander wechselwirken können. Längere nukleosomenfreie Bereiche sind sehr steif und verhindern so Wechselwirkungen von Nukleosomen aus zwei angrenzenden Domänen“, erklärt Kimberly Quililan, die als Masterstudentin maßgeblich an den Experimenten im Labor mitgewirkt hat.
Nicht nur für Hefe relevant
Die Erkenntnisse, die die Forschenden am Modellorganismus der Hefe gewonnen haben, sind auch darüber hinaus relevant: Ähnliche Mechanismen könnten auch bei der 3D-Organisation im menschlichen Genom eine Rolle spielen. Die Ergebnisse des Göttinger Teams könnten zukünftig dazu beitragen, mögliche Ursachen für Krankheiten besser zu erkennen, die auf einer Störung der Genregulation beruhen. „Wenn wir verstehen, wie die Gene zugänglich und dadurch aktivierbar werden, kann dies ein nützlicher Ansatzpunkt auch für die Prävention und Behandlung entsprechender Krankheiten sein“, sagt Oudelaar.
Die Deutsche Forschungsgemeinschaft förderte die Arbeiten der Göttinger Wissenschaftler*innen im Rahmen des Sonderforschungsbereichs „SFB 1565“.
Wissenschaftlicher Ansprechpartner:
Dr. Marieke Oudelaar
Lise-Meitner-Gruppe Genomorganisation und -regulation
Max-Planck-Institut für Multidisziplinäre Naturwissenschaften, Göttingen
Tel.: +49 551 201-2881
E-mail: marieke.oudelaar@mpinat.mpg.de
Dr. Elisa Oberbeckmann
Abteilung Molekularbiologie
Max-Planck-Institut für Multidisziplinäre Naturwissenschaften, Göttingen
Tel.: +49 551 201-2809
E-mail: elisa.oberbeckmann@mpinat.mpg.de
Originalpublikation:
Oberbeckmann, E.; Quililan, K.; Cramer, P.; Oudelaar, A. M.: In vitro reconstitution of chromatin domains shows a role for nucleosome positioning in 3D genome organization. Nature Genetics (30. Januar 2024).
https://doi.org/10.1038/s41588-023-01649-8
Weitere Informationen:
https://www.mpinat.mpg.de/4614736/pr_2404 – Original-Pressemitteilung
https://www.mpinat.mpg.de/de/oudelaar – Webseite der Lise-Meitner-Gruppe Genomorganisation und -regulation, Max-Planck-Institut für Multidisziplinäre Naturwissenschaften, Göttingen
https://www.mpinat.mpg.de/de/cramer – Webseite der Abteilung Molekularbiologie, Max-Planck-Institut für Multidisziplinäre Naturwissenschaften, Göttingen
Die semantisch ähnlichsten Pressemitteilungen im idw