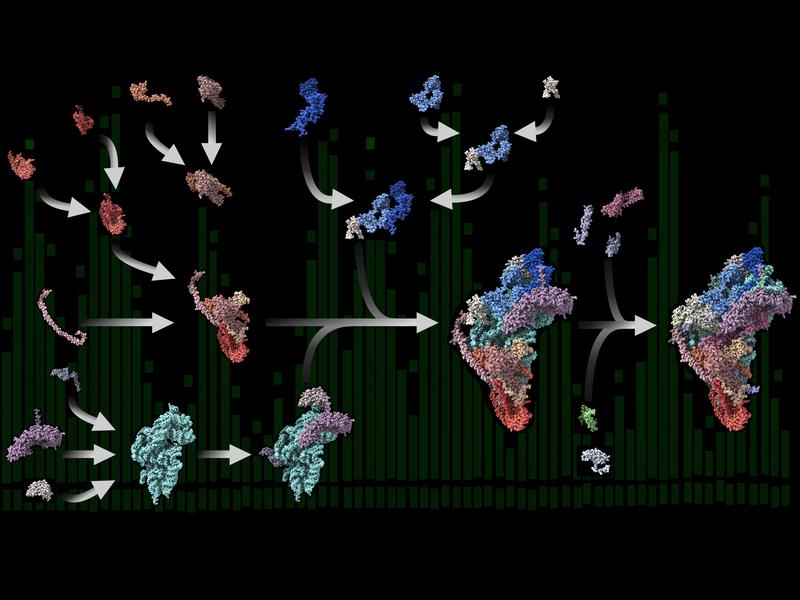
Modulare Bauweise: Neue Erkenntnisse zu Proteinfabriken in menschlichen Mitochondrien
Die „Kraftwerke“ lebender Zellen, die Mitochondrien, sind wahrscheinlich durch Endosymbiose entstanden: Ein Bakterium wanderte in eine Urzelle ein und entwickelte sich schließlich zu einem Organell, das die Zelle unter anderem mit Energie versorgt. Einen Teil der dafür benötigten Proteine stellen Mitochondrien selbst her – mithilfe spezieller Proteinfabriken, den Mitoribosomen. Göttinger Forschende haben nun entschlüsselt, wie die menschliche Zelle die aus Proteinen und RNA bestehenden Mitoribosomen modular zusammensetzt.
Eine Partnerschaft, die vor knapp zwei Milliarden Jahren begann, bestimmt bis heute unser Leben: Eine Urzelle nahm ein freilebendes Bakterium als „Untermieter“ in sich auf. Das Bakterium entwickelte sich im Laufe der Evolution zum Spezialisten für die Energieversorgung: Aufgenommene Nahrung wandelte es mithilfe von Sauerstoff über die sogenannte Atmungskette in nutzbare Energie um. Dafür gab es andere lebenswichtige Funktionen an seine Wirtszelle ab – und trennte sich sogar von einem Teil seines Erbguts, das in das Genom der Wirtszelle integriert wurde. Das einstige Bakterium wurde so zu einem stark spezialisierten Organell. Aus dieser frühen „Lebensgemeinschaft“ entwickelten sich schließlich alle Zellen mit einem Zellkern – und damit alle komplexen Lebewesen bis hin zum Menschen.
Mit der Entwicklung zu höheren Zellen ging auch ein massiver Transfer von Genen einher: Die Mehrheit der DNA aus dem einstigen Bakterien-Genom wurde in das Zellkern-Genom integriert. Nur ein Bruchteil der bakteriellen DNA verblieb im Mitochondrium. Es enthält unter anderem die Bauanleitungen für Schlüsselproteine der Atmungskette.
Fehlende Atmungsketten-Proteine führen zu Krankheiten
Diese Proteine werden daher direkt im Mitochondrium produziert, und zwar von speziellen Proteinfabriken, Mitoribosomen genannt, die nur in diesem Organell vorkommen. Ist die Produktion dieser mitochondrialen Proteinfabriken gestört, führt dies zu einem Mangel an Atmungsketten-Proteinen und in der Folge zu schweren, früh auftretenden Krankheiten.
Die Mitoribosomen zusammenzubauen, ist für die Zelle eine logistische und koordinative Herausforderung, die bislang nur unvollständig untersucht ist: Denn die Bauanleitungen für die Grundbausteine der Mitoribosomen – die ribosomale RNA und 82 ribosomale Proteine – sind in zwei verschiedenen Genomen verschlüsselt. Erstere ist im Mitochondrien-Genom kodiert, letztere sind im Genom des Zellkerns verschlüsselt. Das hat Folgen für das Herstellen der Ribosomen-Proteine. Diese werden mithilfe von Ribosomen im Zytoplasma produziert und dann in die Mitochondrien importiert.
Zusammenbau des menschlichen Mitoribosoms rekonstruiert
Forschungsteams um Ricarda Richter-Dennerlein von der Universitätsmedizin Göttingen (UMG) sowie Juliane Liepe und Henning Urlaub vom Max-Planck-Institut (MPI) für Multidisziplinäre Naturwissenschaften haben jetzt den Zusammenbau des menschlichen Mitoribosoms rekonstruiert – von sehr frühen bis zu späten Schritten. Dieser erfolgt überraschend modular.
„Die Zwischenstufen bei der Bildung der Mitoribosomen sind sehr klein und hochdynamisch. Sie lassen sich daher nur schwer biochemisch untersuchen oder während ihres Zusammenbaus direkt beobachten“, berichtet Richter-Dennerlein, Gruppenleiterin und Professorin an der UMG sowie Mitglied im Exzellenzcluster „Multiscale Bioimaging: Von molekularen Maschinen zu Netzwerken erregbarer Zellen“ (MBExC). Ein multidisziplinärer Ansatz verhalf den Göttinger Teams zum Erfolg. Mithilfe biochemischer Experimente gelang es den Wissenschaftler*innen, Zwischenschritte beim Zusammenbau menschlicher Mitoribosomen zu isolieren und diese zu analysieren. Um den gesamten Prozess Schritt für Schritt zu rekonstruieren, kombinierten sie die so gewonnenen Daten mit Ergebnissen aus Massenspektrometrie-Experimenten, beigetragen durch Urlaubs Gruppe am MPI und an der UMG, und Daten, die das Team um Forschungsgruppenleiterin Liepe vom MPI durch mathematische Modellierungen erhielt. „In einer großartigen Teamleistung konnten wir so schließlich eine fast vollständige Karte des Zusammenbaus der Mitoribosomen erstellen“, sagt Richter-Dennerlein.
Modulare "Bauweise"
Wie die Forschenden herausfanden, werden die im Zytoplasma produzierten Ribosomen-Proteine in die Mitochondrien transportiert und dort zu Modulen zusammengefügt – und zwar in großen Mengen. „Die vorgefertigten Proteinmodule stehen also im Überschuss zur Verfügung“, sagt Liepe. „Nur wenn genügend RNA-Komponenten verfügbar sind, können dann mithilfe spezieller Faktoren die RNA-Bestandteile und Proteinmodule im Organell zu funktionsfähigen Mitoribosomen zusammengesetzt werden.“ Diese bestehen, wie andere Ribosomen auch, aus zwei unterschiedlich großen Untereinheiten.
Elena Lavdovskaia, Erstautorin der jetzt im Fachmagazin Nature Structural & Molecular Biology erschienenen Arbeit, ergänzt: „Die Daten deuten darauf hin, dass das Herstellen der ribosomalen RNA den Zusammenbau der Mitoribosomen begrenzt. Bilden die Mitochondrien zu wenig ribosomale RNA, stoppt der Zusammenbau der Mitoribosomen. Damit haben wir auch eine Erklärung dafür gefunden, wie Mitochondrien den Zusammenbau ihrer Proteinfabriken, die zwei Genome involvieren, koordinieren.“
Von ihren Erkenntnissen erhoffen sich die Forschenden besser zu verstehen, wie der Auf- und Abbau der Proteinfabrik von Mitochondrien erfolgt und wie Störungen hierbei entstehen, die ursächlich für Krankheiten sind.
Wissenschaftlicher Ansprechpartner:
Prof. Dr. Ricarda Richter-Dennerlein
Forschungsgruppe Mitoribosome Assembly
Universitätsmedizin Göttingen
Tel.: +49 551 39-65913
E-Mail: ricarda.richter@med.uni-goettingen.de
Dr. Juliane Liepe
Forschungsgruppe Quantitative und System-Biologie
Max-Planck-Institut für Multidisziplinäre Naturwissenschaften, Göttingen
Tel.: +49 551 201-1471
E-mail: juliane.liepe@mpinat.mpg.de
Prof. Dr. Henning Urlaub
Forschungsgruppe Bioanalytische Massenspektrometrie
Max-Planck-Institut für Multidisziplinäre Naturwissenschaften, Göttingen
Tel.: +49 551 201-1060
E-mail: henning.urlaub@mpinat.mpg.de
Originalpublikation:
Lavdovskaia, E.; Hanitsch, E.; Linden, A.; Pašen, M.; Challa, V.; Horokhovskyi, Y.; Roetschke, H. P.; Nadler, F.; Welp, L.; Steube, E.; Heinrichs, M.; Mai, M. M.-Q.; Urlaub, H.; Liepe, J.; & Richter-Dennerlein, R.: A roadmap for ribosome assembly in human mitochondria. Nat Struct Mol Biol (2024). https://doi.org/10.1038/s41594-024-01356-w
Weitere Informationen:
https://www.mpinat.mpg.de/4743146/pr_2414 – Original-Pressemitteilung
https://biochemie.uni-goettingen.de/index.php/mitoribosome-assembly-ag-richter-dennerlein/ – Forschungsgruppe Mitoribosome Assembly, UMG
https://www.mpinat.mpg.de/de/liepe – Forschungsgruppe Quantitative und System-Biologie, MPI für Multidisziplinäre Naturwissenschaften
https://www.mpinat.mpg.de/de/urlaub – Forschungsgruppe Bioanalytische Massenspektrometrie, MPI für Multidisziplinäre Naturwissenschaften
Die semantisch ähnlichsten Pressemitteilungen im idw