UDE-Forschende nutzen DNA-Origami-Methode: Neue Wege für künstliche Nanofabriken
Künstliche Nanofabriken sind winzige Werkstätten aus körpereigenen Molekülen, die präzise nach Bauplan designt und aufgebaut werden. Sie können künftig beispielweise dazu beitragen, Krankheitsmarker oder Umweltgifte besser zu erkennen oder als hochspezifische Katalysatoren für die Energieumwandlung und -speicherung dienen. Forschende der Universität Duisburg-Essen haben ein Modell entwickelt, das die Entfaltung und den Abbau von Proteinen reguliert und dabei Kompartimentierungsstrategien nutzt – was neue Wege bei der Entwicklung von künstlichen Nanofabriken ebnen könnte. Ihre Ergebnisse wurden nun in Nature Nanotechnology veröffentlicht.
In Zellen sind chemische Reaktionen in kompartimentierten Enzymsystemen organisiert – das heißt, Reaktionen finden in räumlich begrenzten Bereichen, sogenannten Kompartimenten, statt. So werden spezifische Bedingungen für jede Reaktion geschaffen und die Effizienz und Kontrolle der biochemischen Prozesse verbessert. Inspiriert von diesen natürlichen Systemen hat das Forscherteam an der Universität Duisburg-Essen (UDE) unter der Leitung von Prof. Dr. Barbara Saccà vom Zentrum für Medizinische Biotechnologie (ZMB) in Zusammenarbeit mit dem Labor von Prof. Dr. Hemmo Meyer eine künstliche Struktur gebaut, die in verschiedene Bereiche unterteilt ist und spezifische Reaktionen vermittelt. Dafür verwenden sie die DNA-Origami-Methode – eine Technik, bei der DNA-Moleküle wie winzige Bausteine gefaltet werden, um sehr kleine Strukturen zu formen. „Da die Wechselwirkungen zwischen den DNA-Bestandteilen genau bekannt sind, können wir die Struktur, in die sich der Strang faltet, präzise designen – auf Nanometer genau“, erklärt die studierte Chemikerin.
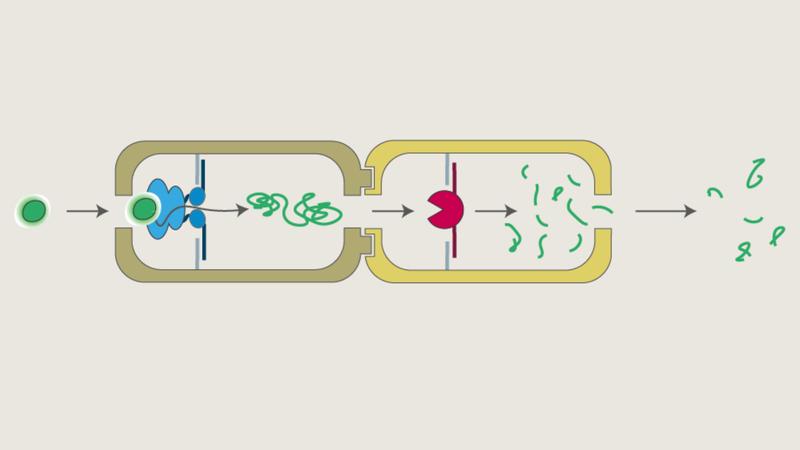
In dem Modell der UDE-Wissenschaftler:innen besteht die künstliche Nanofabrik aus zwei solcher Strukturen, die je einen Hohlraum umschließen. Im ersten Teil ist die Proteinentfaltungsmaschine p97 fest verankert, die – wie ihr Name besagt – Proteine entfaltet. In seiner nun länglichen Form wird das Protein anschließend kontrolliert in den zweiten Bereich transportiert. Dort ist eine Protease gebunden, die den Proteinstrang in kleine Fragmente zerlegt. Durch die räumliche Verbindung der beiden Teilprozesse wird die Reaktion deutlich effizienter: die Reaktionsgeschwindigkeit steigt um das Zehnfache, unerwünschte Nebenreaktionen werden um fast das Sechsfache reduziert im Vergleich zu zwei auseinanderliegenden Reaktionen, die zudem räumlich nicht abgeschottet sind.
„Unsere Ergebnisse zeigen das Potenzial der DNA-Nanotechnologie zur Programmierung modularer und kompartimentierter Enzymsysteme und eröffnen neue Wege für die synthetische Biologie und biokatalytische Anwendungen", sagt Saccà. Für die Zukunft plant die Professorin für Bionanotechnologie, die Origami-Fabriken weiter zu verbessern: zum Beispiel (1) durch die Beschichtung des Origami mit organischen oder anorganischen Materialien, die das Konzept einer semipermeablen Membran nachahmen oder (2) indem der Zugang zur Reaktionskammer nur noch an einer Stelle möglich wird und so die Prozesse im Innern besser gegen äußere Einflüsse geschützt sind. „Fortschritte dieser Art könnten zur Entwicklung von Miniaturlaboratorien führen, deren Fähigkeiten über die natürlichen Systeme hinausgehen.“
Im Bild:
Das Substrat (grün) wird von der Entfaltungsmaschine p97 (blau) erkannt, die in einem ersten Nanokompartiment in eine eindeutige und genau definierte Richtung ausgerichtet ist. Das Substrat wird dann entfaltet und in ein zweites Kompartiment verlagert, in dem sich eine Protease (rot) befindet, die die Aufspaltung des Substrats in kleinere Peptidfragmente katalysiert.
Wissenschaftlicher Ansprechpartner:
Prof. Dr. Barbara Saccà, Zentrum für Medizinische Biotechnologie, Tel. 0201/183-3395, barbara.sacca@uni-due.de
Originalpublikation:
https://www.nature.com/articles/s41565-024-01738-7
Die semantisch ähnlichsten Pressemitteilungen im idw