Die Rechnung ohne den Wirt gemacht: Übertragung der Tuberkulose hängt nicht nur vom Erreger ab
Weltweit existieren verschiedene Gruppen von TB-Bakterien mit unterschiedlicher regionaler Ausbreitung: Manche sind Generalisten und auf vielen Kontinenten zu finden, andere sind in ihrer Ausbreitung sehr begrenzt. Ein internationales Forscherteam konnte nun erstmals zeigen, dass die Spezialisten-Stämme sich effektiver unter passenden Wirten aus demselben geografischen Gebiet verbreiten, wogegen Generalisten Stämme sich in verschiedenen Wirtspopulationen ausbreiten können. Die Übertragbarkeit der Tuberkulose hängt somit nicht nur vom Erreger oder Wirt, sondern auch von deren Kombination ab.
Tuberkulose zählt mit über 1,4 Millionen Todesfällen pro Jahr immer noch zu den gefährlichsten Infektionskrankheiten weltweit. Insgesamt kommen auf der Welt zehn unterschiedliche genetische Linien des Erregers, dem Bakterium aus dem Mycobacterium tuberculosis-Komplex (Mtbc), vor: In Europa und Nordamerika sind Stämme der Linie L4 am häufigsten verbreitet, während im Asiatischen Raum Stämme der Linie L2 vorherrschend sind. Afrika ist der einzige Ort wo regelmäßig Stämme der Linien 5 und 6 gefunden werden. Während Stämme der Linien L2 und L4 weit verbreitet sind und häufig vorkommen, wurden Stämme einiger Linien des afrikanischen Raums nur selten außerhalb des Kontinents isoliert und weisen eine begrenzte geografische Verbreitung auf.
Es wird daher angenommen, dass sich geografisch begrenzte Linien des Mtbc sich effektiver unter sympatrischen (passenden) Wirten, d. h. solchen, die sich im selben geografischen Gebiet aufhalten, verbreiten und eine geringere Übertragbarkeit bei allopatrische (nicht passenden) Wirten, also auf Menschen aus unterschiedlichen geographischen Regionen, aufweisen. Bislang konnte diese Annahme jedoch nicht bewiesen werden, da es keine großen Datensätze mit klinischen Tuberkulosedaten und hochauflösenden Pathogensequenzen gab.
Nun ist es einem internationalen Team aus Wissenschaftlerinnen und Wissenschaftlern des Forschungszentrums Borstel, Leibniz Lungenzentrum, der Charité – Universitätsmedizin Berlin und der Harvard Medical School in Boston erstmal gelungen, diese Hypothese zu belegen: Anhand von Erregergenom- und Kontaktverfolgungsdaten von 2.279 Tuberkulosefällen, die mit 12.749 sozialen Kontakten aus drei Städten mit geringer Inzidenz verbunden sind, konnten die Forschenden zeigen, dass Stämme geografisch begrenzte Mtbc-Linien weniger übertragbar sind als Stämme von Linien, die eine weite globale Verbreitung aufweisen. Die Daten stammen aus Hamburg, New York und Amsterdam und wurden in Zusammenarbeit mit den jeweiligen Gesundheitsämtern zusammengestellt. Das Team konnte zeigen, dass bei allopatrischer Erreger-Wirt-Exposition, bei der der Erreger und der Wirt aus sich nicht überschneidenden Gebieten stammen, die Wahrscheinlichkeit einer Infektion bei den Kontaktpersonen um 38 % geringer war als bei sympatrischer Exposition.
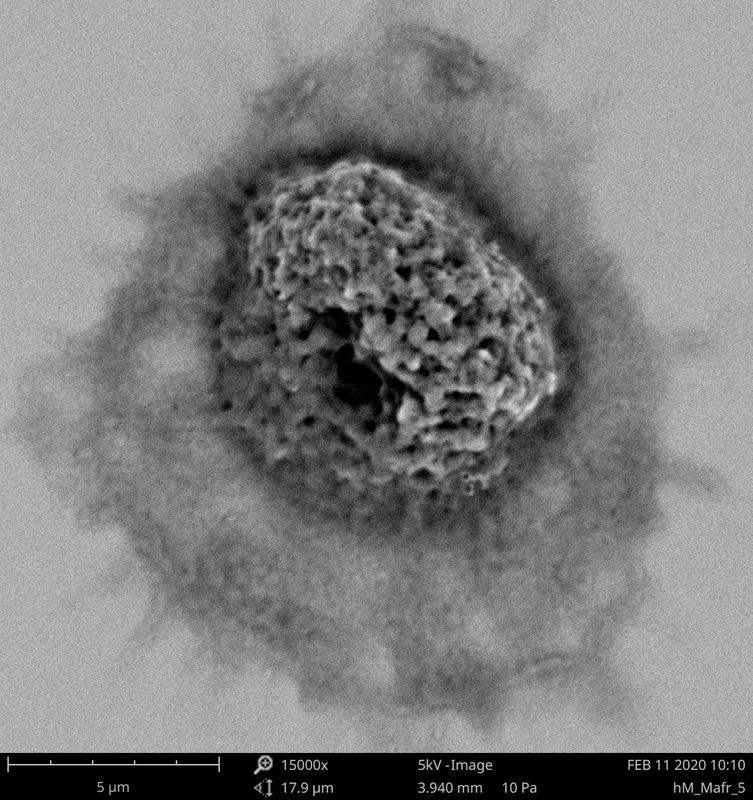
Diese epidemiologischen Beobachtungen wurden durch Laborexperimente unterstützt. Mit Hilfe eines Makrophagen-Infektionsmodells konnte gezeigt werden, dass eine geringere Aufnahme und ein geringeres Wachstum von Mtbc in allopatrischen Makrophagen nach der ersten Exposition festgestellt werden konnte. Diese Untersuchungen unterstützen die Resultate der epidemiologischen Analysen und werden in zukünftigen Projekten noch um weitere Wirts-Stamm Kombinationen erweitert.
Die Forschenden kommen zu dem Schluss, dass die langfristige Koexistenz von Mtbc-Stämmen und Menschen zu einer unterschiedlichen Übertragbarkeit von Mtbc-Stämmen geführt hat und diese je nach menschlicher Population unterschiedlich ist. „Diese Unterschiede in der Übertragbarkeit führen dazu, dass Stämme geografisch begrenzter Mtbc-Linien eine Barriere für ihre Fähigkeit haben, sich in andere geografische Regionen auszubreiten,“ so Dr. Dr. Matthias Gröschel, Erstautor der Studie und Arzt im Fächerverbund Infektiologie, Pneumologie und Intensivmedizin der Charité. „Wir wollen in anschließenden Arbeiten nun darauf abzielen, die molekularen Grundlagen allopatrischer und sympatrischer Wirt -Pathogen Interaktion zu verstehen.“
Diese Informationen können in Zukunft hilfreich sein, um Umgebungsuntersuchungen zu personalisieren und beispielsweise Hochrisiko-Kontakte wie sympatrische Wirts-Pathogen Kontakte mit höherer Priorität nachzuverfolgen. Auch bei der Entwicklung von Medikamenten könnten diese spezifische Virulenzmechanismen von Bedeutung sein.
„Die Arbeiten erlauben erstmals ein genaueres Verständnis der Wirt-Pathogen Interaktion bei der Tuberkulose auf globalem Niveau“ sagt Prof. Stefan Niemann, Leiter der Studie am Forschungszentrum Borstel, Leibniz Lungenzentrum. „Wir arbeiten mit Hochdruck daran, die pathobiologischen Mechanismen aufzuklären“.
Diese Arbeit wurde gefördert durch das National Institutes of Health, die Deutsche Forschungsgemeinschaft (DFG), das BIH Charité Junior Digital Clinician Scientist Programm der Charité und des Berlin Institute of Health in der Charité (BIH), dem Leibniz-Wissenschaftscampus EvoLUNG (Evolutionäre Medizin der Lunge), den Exzellenzcluster „Precision Medicine in Chronic Inflammation“ (PMI) und das Deutsche Zentrum für Infektionsforschung (DZIF).
Wissenschaftlicher Ansprechpartner:
Dr. Dr. Matthias Gröschel
Fächerverbund für Infektiologie, Pneumologie und Intensivmedizin
Campus Charité Mitte (CCM) und Campus Virchow-Klinikum (CVK)
Charité – Universitätsmedizin Berlin
Charitéplatz 1, 10117 Berlin
E-Mail: matthias.groeschel@charite.de
Prof. Dr. Stefan Niemann
Programmdirektor Programmbereich Infektionen
Forschungszentrum Borstel, Leibniz Lungenzentrum
Parkallee 1, 23845 Borstel
Email: sniemann@fz-borstel.de
Originalpublikation:
Matthias I. Gröschel, Francy J. Pérez-Llanos, Roland Diel, Roger Vargas Jr, Vincent Escuyer, Kimberlee Musser, Lisa Trieu, Jeanne Sullivan Meissner, Jillian Knorr, Don Klinkenberg, Peter Kouw, Susanne Homolka, Wojciech Samek, Barun Mathema, Dick van Soolingen, Stefan Niemann, Shama Desai Ahuja, & Maha R. Farhat. Differential rates of Mycobacterium tuberculosis transmission associate with host–pathogen sympatry. Nature Microbiology 2024 DOI: 10.1038/s41564-024-01758-y
Ähnliche Pressemitteilungen im idw