Maßgeschneiderte Mikroben-Gemeinschaften
Biologie: Veröffentlichung in Synthetic Biology
Wie können Computermodelle dabei helfen, mikrobielle Gemeinschaften zu designen? Im Rahmen des von der Heinrich-Heine-Universität Düsseldorf (HHU) koordinierten Sonderforschungsbereichs SFB1535 „MibiNet“ untersuchte ein Forschungsteam aus Aachen, Düsseldorf und East Lansing / USA die Entwicklungsperspektiven der sogenannten Synthetischen Biologie. In der Fachzeitschrift Synthetic Biology erläutern sie, warum sie der rechnergestützten Biologie dabei eine besondere Rolle beimessen.
Gemeinschaften von Mikroorganismen – Bakterien, Pilzen und Viren – finden sich überall, auch und gerade in Organismen. Sie erfüllen dort unterschiedliche Funktionen. So ist die mikrobielle Gemeinschaft im menschlichen Darm, das sogenannte Mikrobiom, essentiell für den Stoffwechsel; erst die Mikroorganismen schließen viele Nährstoffe auf und machen sie dem Körper verfügbar. Ist das Mikrobiom falsch zusammengesetzt, kann dies dem Gesamtorganismus erheblich schaden.
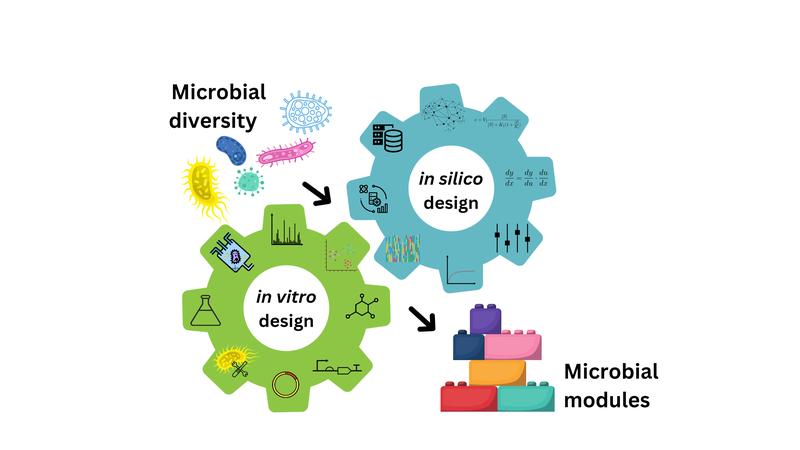
Mit diesen mikrobiellen Netzwerken befasst sich zunehmend auch das interdisziplinäre Forschungsfeld der „Synthetischen Biologie“. Deren Ziel ist es, mithilfe ingenieurwissenschaftlicher Prinzipien neue biologische Systeme und Organismen zu entwerfen und zu bauen, die spezifische Funktionen erfüllen können. Gentechnische Methoden helfen dabei, DNA und RNA zu verändern und zwischen verschiedenen Organismen zu übertragen. Zunächst fokussierte sich die Synthetische Biologie auf einzelne synthetische Organsimen, inzwischen wird aber ihr Potenzial für das Design hochkomplexer Netzwerke, wie z.B. künstliche Gemeinschaften aus (synthetischen) Organismen, augenfällig.
Mögliche Anwendungsgebiete solcher künstlichen Gemeinschaften sind breit gefächert. Sie umfassen beispielsweise die Eindämmung von Krankheiten, die Produktivitätssteigerung von Nutzpflanzen oder die Produktion wertvoller Biomoleküle.
Forschende des SFB1535 MibiNet haben sich von natürlichen Flechten inspirieren lassen, in denen phototrophe Cyanobakterien oder Algen in enger symbiotischer Wechselwirkung mit heterotrophen Pilzpartnern stehen. Sie wollen die hier beispielhaft manifestierte mikrobiellen Vernetzung für künftige Anwendungen erschließen. Die Forschungsergebnisse sollen etwa dazu beitragen, interdisziplinäre Methoden und Technologien für CO2-negative Prozesse – also solche, die aktiv CO2 aus der Atmosphäre ziehen und fixieren – zu schaffen. In einem weiteren Forschungsprojekt ACCeSS soll die Sonnenenergie nutzbar gemacht werden, um Abwasser zu behandeln.
In der Fachzeitschrift Synthetic Biology umreißen Forschende der RWTH Aachen, der HHU und der Michigan State University (MSU) in East Lansing in den USA diese zukünftige Entwicklungslinie der Synthetischen Biologie. Sie betonen darin die Rolle der computergestützten Biologie als integraler Baustein, die das Design künstlicher Gemeinschaften erheblich vereinfachen kann.
Prof. Dr. Ilka Axmann von der HHU, Korrespondenzautorin der Studie: „Wir schlagen einen Perspektivwechsel vor, weg von einzelnen Organismen als solche hin zu den funktionalen Beiträgen, die Organismen innerhalb der Gemeinschaft leisten.“ Sie ergänzt zum Forschungsansatz: „Im Fokus steht die Funktion, die die Gemeinschaft insgesamt erfüllen soll. Es ist dafür unerheblich, welche speziellen Organismen sie enthält. Diese sind lediglich das Chassis, das die notwendigen Stoffwechselwege enthält und bereitstellt, um die erforderlichen funktionellen Aufgaben zu erledigen.“
Dr. Daniel C. Ducat, Professor für Biochemie und Molekularbiologie an der MSU, ergänzt: „Es gibt immer mehr Beispiele dafür, dass sich zwar die spezifische Artenzusammensetzung komplexer mikrobieller Gemeinschaften im Laufe der Zeit oder an anderen Orten ändern kann; die spezifischen Funktionen der Gemeinschaft sind jedoch in einem größeren Maßstab stabil.“
Dr. Anna Matuszyńska, Erstautorin der Studie und Juniorprofessorin für Computational Life Science an der RWTH: „Die computergestützte Biologie kann dabei helfen, eine wünschenswerte Modularisierung in der Synthetischen Biologie zu unterstützen, die sowohl die Komplexität reduziert als auch vielseitige, skalierbare Rahmenwerke schafft, die auf bestimmte Funktionen innerhalb biologischer Gemeinschaften zugeschnitten werden können. Mithilfe mathematischer Modelle können wir solche Systeme vorhersagen und optimieren, damit sie zuverlässig und effizient arbeiten. Dieses ‚In-silico-Design‘ sollte bereits in den frühesten Stadien des Aufbaus einer synthetischen Gemeinschaft eingesetzt werden."
Originalpublikation:
Anna Matuszyńska, Oliver Ebenhöh, Matias D. Zurbriggen, Daniel C. Ducat, Ilka M. Axmann. A new era of synthetic biology – microbial community design. Synthetic Biology, 2024, 9(1), ysae011.
DOI: 10.1093/synbio/ysae011
Weitere Informationen:
https://www.sfb1535.hhu.de/mibinet
https://www.access.hhu.de/
Ähnliche Pressemitteilungen im idw