Forschende simulieren mit KI visuelles System im Gehirn der Fruchtfliege
Internationalem Forscherteam aus Tübingen und Virginia gelingt durch künstliches neuronales Netz erstmals Vorhersage der Berechnungen eines lebenden Gehirns.
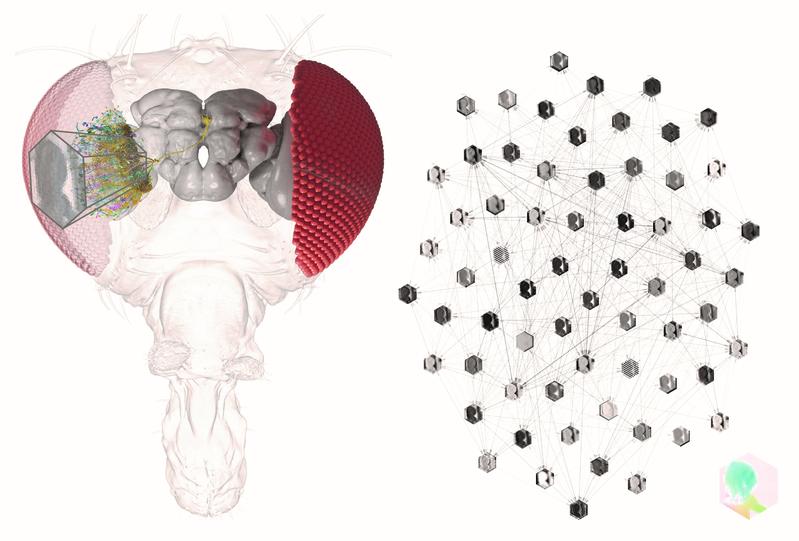
Informationen im Gehirn werden über elektrische Signale zwischen spezialisierten Zellen, den Neuronen, übertragen. Große Netzwerke solcher Neuronen steuern Wahrnehmungen, Verhalten und Kognition. Die Wissenschaft hat lange nach Möglichkeiten gesucht, neuronale Netze im Gehirn mit Computern zu simulieren, um zu verstehen, wie sie funktionieren. Mit neuen Erkenntnissen über die neuronalen Schaltpläne im Gehirn der Fruchtfliege und Methoden der künstlichen Intelligenz gelang es nun Forschenden, ein neuronales Netz zu knüpfen, welches zuvor kaum Vorstellbares leistet: Es sagt die Aktivität einzelner Neuronen vorher, ohne dass Messungen an einem lebenden Gehirn vorgenommen werden müssen. Die Studie von Professor Jakob Macke und Dr. Janne Lappalainen von der Universität Tübingen und Dr. Srinivas Turaga und Kollegen vom Janelia Research Campus des Howard Hughes Medical Institute in Ashburn, Virginia (USA), ist am Mittwoch in der Fachzeitschrift nature erschienen.
Seit Jahrzehnten messen Neurowissenschaftlerinnen und -wissenschaftler neuronale Aktivitäten lebender Tiere, weil sie die Zusammenhänge zwischen Gehirnaktivität und Verhalten besser verstehen wollen. Diese Experimente haben bahnbrechende Erkenntnisse über die Funktionsweise des Gehirns erbracht - aber ein Großteil des Gehirns ist nach wie vor unerforscht.
Die Teams aus Tübingen und Virginia haben nun mithilfe von künstlicher Intelligenz und des Konnektoms, einer Karte der Neuronen und ihrer im Hirngewebe bestehenden Verbindungen, die Aktivität der Neuronen im lebenden Gehirn vorhergesagt. Indem sie lediglich Informationen aus dem Konnektom des visuellen Systems der Fruchtfliege verwenden sowie Annahmen über die Funktionen des Schaltkreises machen, haben die Forscher eine KI-Simulation erstellt, die die Aktivität jedes Neurons im Schaltkreis vorhersagen kann. "Wir verfügen nun über eine Berechnungsmethode, mit der wir Messungen des Konnektoms in Vorhersagen über die neuronale Aktivität und die Gehirnfunktion umsetzen können, ohne zuerst aufwändige Messungen am lebenden Neuron durchführen zu müssen", sagt Srinivas Turaga, Leiter der Janelia-Forschungsgruppe und einer der Hauptautoren der neuen Studie.
Das Forschungsteam nutzte das Konnektom, um eine detaillierte mechanistische Netzwerksimulation des visuellen Systems der Fliege zu erstellen, bei der jedes modellierte Neuron einem realen Neuron und jede modellierte Synapse einer realen Synapse im Gehirn entspricht. Obwohl sie die Dynamik der Neuronen im realen Gewebe nicht kannten, konnte das Team diese unbekannten Parameter mit Hilfe von Deep-Learning-Methoden vorhersagen. Dazu verbanden sie die Informationen aus dem Konnektom mit ihrem Wissen über die Funktion des Schaltkreises: Dem Erkennen von Bewegungen. "Mit dieser Kombination konnten wir prüfen, ob unser auf dem Konnektom basierende Ansatz ein gutes Modell des Gehirns liefern kann“, sagt Janne Lappalainen, Doktorand an der Universität Tübingen und Erstautor der Studie.
Das neue Modell sagt die Aktivität von 64 verschiedenen Neuronentypen des visuellen Systems der Fruchtfliege voraus und reproduziert die Ergebnisse aus über zwei Dutzend experimentellen Studien der letzten zwei Jahrzehnte. Laut den Autoren hat die neue Arbeit das Potenzial, die Hirnforschung grundlegend zu verändern, indem Vorhersagen über die Aktivität einzelner Neurone nun direkt aus dem Konnektom abgeleitet werden können. Im Prinzip kann man mit dem Modell jedes beliebige Experiment simulieren, so dass daraus abgeleitete Vorhersagen dann im Labor getestet werden können.
Die neue Forschungsarbeit enthält über 450 Seiten mit aus dem Modell abgeleiteten Vorhersagen, einschließlich der Identifizierung von Zellen, von denen bisher nicht bekannt war, ob sie an der Bewegungserkennung beteiligt sind, und die nun in Fruchtfliegen untersucht werden können. "Bislang bestand eine große Lücke zwischen dem statischen Konnektom und der Dynamik der Berechnungen im lebenden Gehirn. Die Frage war, ob wir diese Lücke mit einem Modell schließen können. Am konkreten Beispiel der Fruchtfliege ist uns dies nun gelungen", sagt Jakob Macke, einer der Hauptautoren der Studie. Mit diesem Ansatz ist es möglich, künstliche neuronale Netze zu schaffen, die dem Gehirn der Fruchtfliege ähnlich sind und künftig für eine Vielzahl von Untersuchungen genutzt werden können: Zum Beispiel könnte mit ihnen untersucht werden, warum biologische neuronale Netze um Größenordnungen effizienter sind als künstliche neuronale Netze.
Janne Lappalainen ist Doktorand an der Universität Tübingen und der International Max Planck Research School "Intelligente Systeme" und Gastwissenschaftler am Janelia Research Campus des HHMI. Professor Jakob Macke leitet die Gruppe "Maschinelles Lernen in der Wissenschaft", als Teil des Tübinger Exzellenzclusters "Maschinelles Lernen: Neue Perspektiven für die Wissenschaft” und ist Forscher am Tübingen AI Center sowie dem "Bernstein Center for Computational Neuroscience Tübingen". Das Projekt wurde zum Teil durch den ERC Consolidator Grant DeepCoMechTome finanziert.
Wissenschaftlicher Ansprechpartner:
Prof. Dr. Jakob Macke
Universität Tübingen
Exzellenzcluster „Maschinelles Lernen in der Wissenschaft“
jakob.macke@uni-tuebingen.de
Originalpublikation:
Janne K. Lappalainen, Fabian D. Tschopp, Sridhama Prakhya, Mason McGill, Aljoscha Nern, Kazunori Shinomiya, Shinya Takemura, Eyal Gruntman, Jakob H. Macke, Srinivas C. Turaga: "Connectome-constrained networks predict neural activity across the fly visual system", Nature, 2024, https://www.nature.com/articles/s41586-024-07939-3, DOI 10.1038/s41586-024-07939-3.
Weitere Informationen:
http://Research Briefing:
http://doi.org/10.1038/d41586-024-02935-z
Ähnliche Pressemitteilungen im idw