Bessere MRT-Bewegtbilder durch neue Machine-Learning-Methode
Forschenden der TU Graz gelingt es mithilfe raffiniert trainierter neuronaler Netze, aus nur wenigen MRT-Messdaten präzise Echtzeitbilder des schlagenden Herzens zu erzeugen. Das Verfahren kann auch andere MRT-Anwendungen beschleunigen.
Die medizinische Bildgebung mittels Magnetresonanztomographie (MRT) ist sehr zeitaufwändig, da ein Bild aus Daten von vielen Einzelmessungen zusammengesetzt werden muss. Durch den Einsatz von Machine Learning gelingt die Bildgebung auch mit weniger MRT-Messdaten, was Zeit und Kosten spart. Die Voraussetzung dafür aber sind perfekte Bilder, um damit die KI-Modelle zu trainieren. Für gewisse Anwendungen wie z.B. Echtzeit-Bewegtbild-MRT gibt es solche perfekten Trainingsbilder nicht, da solche Aufnahmen bisher immer etwas unscharf sind. Einem internationalen Forschungsteam um Martin Uecker und Moritz Blumenthal vom Institute of Biomedical Imaging an der TU Graz ist es nun mithilfe raffiniert trainierter neuronaler Netze gelungen, präzise Live-MRT-Bilder des schlagenden Herzens auch ohne solche Trainingsbilder und mit nur sehr wenigen MRT-Daten zu erzeugen. Durch diese Verbesserungen könnte die Echtzeit-Bewegtbild-MRT zukünftig häufiger in der Praxis zum Einsatz kommen.
Kalibrierung der Bildgebung durch vorenthaltene Daten
Um ihr Machine-Learning-Modell für die MRT-Bildgebung zu trainieren, nutzten Martin Uecker und Moritz Blumenthal Methoden des „Self-supervised Learnings“: Dabei sind nicht vorab kuratierte perfekte Bilder die Grundlage zum Trainieren des Modells, sondern eine Teilmenge der Ausgangsdaten, aus denen das Modell die Bilder rekonstruieren soll. Moritz Blumenthal erklärt es so: „Wir haben die vom MRT-Gerät gelieferten Messdaten in zwei Portionen aufgeteilt. Aus der ersten, größeren Datenportion rekonstruiert unser Machine-Learning-Modell das Bild. Anschließend versucht es, auf Basis des Bildes die ihm vorenthaltene, zweite Portion der Messdaten zu berechnen.“ Gelingt dem System dies nicht oder nur schlecht – so die zugrundeliegende Logik –, muss das zuvor rekonstruierte Bild falsch gewesen sein. Das Modell wird aktualisiert, es erstellt eine neue verbesserte Bildvariante und versucht erneut, die zweite Datenportion zu berechnen. Dieser Vorgang läuft über eine Vielzahl von Runden, bis das Ergebnis stimmig ist. Dabei lernt das System aus einer Vielzahl solcher Rekonstruktionen in diesem Trainingsprozess, wie gute MRT-Bilder aussehen sollten. Später, während der Anwendung, kann das Modell dann direkt ein gutes Bild berechnen.
Verfahren kann viele MRT-Anwendungen schneller und günstiger machen
„Unser Verfahren ist anwendungsreif“, sagt Martin Uecker, „auch wenn es vermutlich noch etwas dauern wird, bis es in der Praxis auch tatsächlich eingesetzt wird.“ Die Methode kann für viele weitere MRT-Anwendungen verwendet werden, um diese schneller und damit günstiger zu machen. Dazu zählt etwa die quantitative MRT, bei der physikalische Gewebe-Parameter exakt gemessen und quantifiziert werden. „Dadurch können Radiolog*innen für Diagnosen auf exakte Daten zurückgreifen, anstatt Bilder anhand von Helligkeitsunterschieden auf Basis von Erfahrungswerten interpretieren zu müssen“, erläutert Martin Uecker. „Bislang dauern quantitative MRT-Messungen aber oft sehr lang. Mit unserem Machine-Learning-Modell konnten wir diese Messungen ohne Qualitätseinbußen stark beschleunigen.“
Die Forschungsergebnisse, die kürzlich im Fachmagazin Magnetic Resonance in Medicine veröffentlicht wurden, sind das Ergebnis einer internationalen und interdisziplinären Kooperation des Institutes of Biomedical Imaging: Beteiligt waren u.a. Christina Unterberg (Kardiologin an der Universitätsmedizin Göttingen), Markus Haltmeier (Mathematiker an der Universität Innsbruck), Xiaoqing Wang (MRT-Forscher an der Harvard Medical School) und Chiara Fantinato (Erasmusstudentin aus Italien). Die Algorithmen und MRT-Daten sind öffentlich, sodass andere Forschende die Ergebnisse direkt nachvollziehen und auf der neuen Methode aufbauen können.
MRT-Video eines schlagenden Herzens zum Download:
https://cloud.tugraz.at/index.php/s/iJZeHxHgfNGJYHb
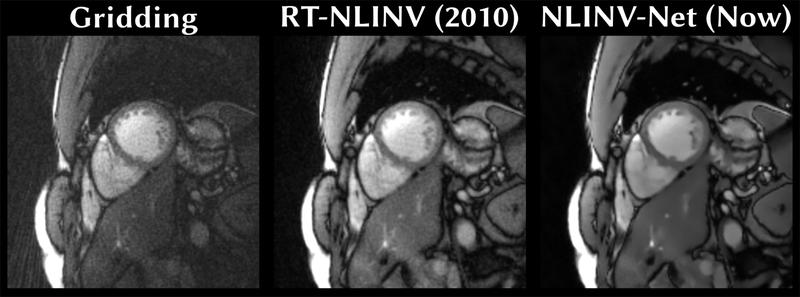
(Kurzachsenschnitt durch das Herz: Gegenüberstellung der einfachen Bildrekonstruktionsmethode "Gridding" [links], der 2010 entwickelten Methode "RT-NLINV" [Mitte] sowie der neuen Methode "NLINV-Net" [rechts], bei der die Modellierung von RT-NLINV mit maschinellem Lernen kombiniert wird, was eine wesentlich schnellere und weiter verbesserte Bildrekonstruktion ermöglicht.)
Wissenschaftlicher Ansprechpartner:
Martin UECKER
Univ.-Prof. Dipl.-Phys. Dr.rer.nat.
TU Graz | Institute of Biomedical Imaging
Tel.: +43 316 873 35400
uecker@tugraz.at
Originalpublikation:
Self-supervised learning for improved calibrationless radial MRI with NLINV-Net
Autor*innen: Moritz Blumenthal, Chiara Fantinato, Christina Unterberg-Buchwald, Markus Haltmeier, Xiaoqing Wang, Martin Uecker
In: Magnetic Resonance in Medicine 2024.
https://onlinelibrary.wiley.com/doi/10.1002/mrm.30234
DOI: 10.1002/mrm.30234
Weitere Informationen:
https://www.tugraz.at/forschung/fields-of-expertise/information-communication-computing/ueberblick-information-communication-computing Diese Forschung ist im Field of Expertise „Information, Communication & Computing“ verankert, einem von fünf strategischen Schwerpunktfeldern der TU Graz.
Die semantisch ähnlichsten Pressemitteilungen im idw