ONE-Mikroskopie: mit dem Lichtmikroskop vom Molekül zur 3D-Struktur
Forschende der Universitätsmedizin Göttingen haben ein neues Verfahren entwickelt, das es erstmals erlaubt, die 3D-Form von Proteinen mit einem herkömmlichen Mikroskop abzubilden. Die mit Künstlicher Intelligenz kombinierte One-step Nanoscale Expansion (ONE)-Mikroskopie ermöglicht die Erkennung von Strukturveränderungen geschädigter oder toxischer Proteine in menschlichen Proben. Erkrankungen wie Parkinson, die auf Proteinfehlfaltungen beruhen, könnten somit frühzeitig erkannt und behandelt werden. Die ONE-Mikroskopie wurde von der Zeitschrift Nature als eine der „sieben Technologien, die man 2024 im Auge behalten sollte“ bezeichnet, und kürzlich in Nature Biotechnology veröffentlicht.
Die Fluoreszenz-Bildgebung ist eines der vielseitigsten und am weitesten verbreiteten Instrumente in der Biologie, um biologische Prozesse in lebenden Zellen zu beobachten. Trotz der Fortschritte in der Technik und der Verbesserung der Auflösung bleibt die Darstellung einzelner Moleküle und die Organisation molekularer Komplexe mittels Fluoreszenzmikroskopie eine Herausforderung. Dies war bisher ausschließlich mit teuren strukturbiologischen Methoden, wie der Elektronenmikroskopie (EM) und insbesondere der Kryo-EM möglich, bei der die Proben mit einem sehr starken Elektronenstrahl bei extrem niedriger Temperatur abgebildet werden.
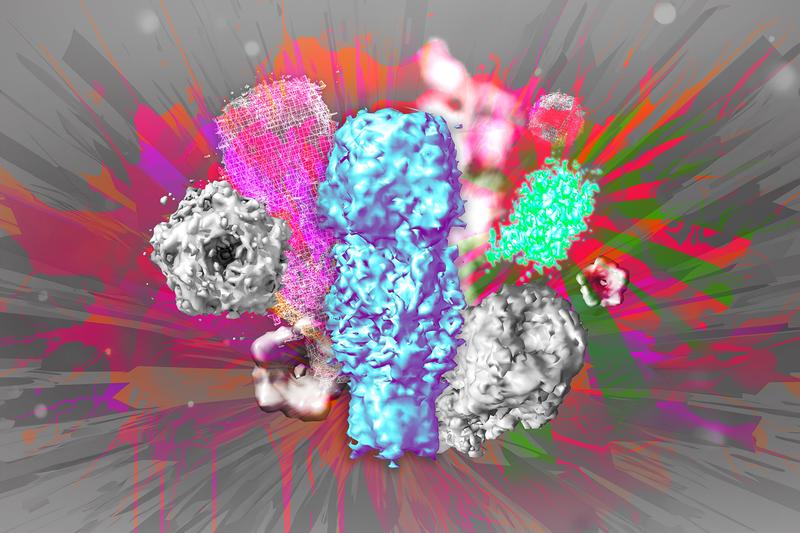
Ein Forschungsteam unter der Leitung von Prof. Dr. Silvio O. Rizzoli, Direktor der Instituts für Neuro- und Sinnesphysiologie der Universitätsmedizin Göttingen (UMG), Sprecher des Center for Biostructural Imaging of Neurodegeneration (BIN) sowie Mitglied des Exzellenzclusters „Multiscale Bioimaging: von molekularen Maschinen zu Netzwerken erregbarer Zellen“ (MBExC), und Dr. Ali Shaib, Gruppenleiter im Institut für Neuro- und Sinnesphysiologie der UMG, hat nun mit ein paar einfachen, aber effektiven Tricks eine Methode entwickelt, um einzelne Moleküle mit herkömmlicher Lichtmikroskopie detailgenau darzustellen. Statt teure, hochauflösende Mikroskope einzusetzen, um die Auflösung zu verbessern, entwickelten sie die Einstufige Nanoskalen-Expansions (One-step Nanoscale Expansion; ONE)-Mikroskopie. Bei diesem Verfahren wird das Volumen der Probe vergrößert, indem die Zellen und die darin enthaltenen Strukturen auf ein wasserabsorbierendes Gel gebunden werden, welches in die Zellen eindringt. Durch die Aufnahme von Wasser vergrößert sich das Gel bis auf das 15-fache seines Volumens. Die Moleküle in der Probe bewegen sich dadurch gleichmäßig auseinander und werden ebenfalls größer, so dass sie nach gezielter Markierung mit fluoreszierenden Molekülen mit einem Lichtmikroskop abgebildet werden können. Kombiniert mit einem auf Künstlicher Intelligenz basierenden Verfahren zur Auswertung der Fluoreszenzveränderungen, gelang den Wissenschaftler*innen erstmals, was bisher nur mit der hochauflösenden Kryo-Elektronenmikroskopie und Röntgentechnologie möglich war: „Wir sind jetzt in der Lage, 3D-Proteinstrukturen aus zweidimensionalen Fluoreszenzbildern zu rekonstruieren,“ so Prof. Rizzoli. Dies bietet eine noch nie dagewesene Möglichkeit, feine strukturelle Details einzelner Proteine sowie von Multiproteinkomplexen in Zellen oder auch isoliert direkt darzustellen. Auch Veränderungen in der räumlichen Struktur von Proteinen sind mit der ONE-Mikroskopie leicht aufzudecken. In einer Kooperation mit Kolleg*innen aus Göttingen und Kassel konnten molekulare Proteinaggregate, die typisch für die Parkinson-Erkrankung sind, in Hirnwasser-Proben von Patient*innen abgebildet und klassifiziert werden. Das ist vielversprechend für eine verbesserte Früherkennung der Parkinson-Erkrankung, von der weltweit Millionen Menschen betroffen sind.
Die ONE-Mikroskopie ist ein einfaches und kostengünstiges Verfahren, das in jedem Labor mit einem herkömmlichen Mikroskop durchgeführt werden kann und ein Auflösungsniveau im Bereich von zirka ein Nanometer erreicht. Das ist etwa 100.000 Mal kleiner als der Durchmesser eines menschlichen Haares. Die benötigte Software stellen die Autoren als kostenloses Open-Source-Paket zur Verfügung. Das neu entwickelte Verfahren wurde jetzt in der renommierten Fachzeitschrift Nature Biotechnology veröffentlicht. Aufgrund des hohen Potentials hat Nature die ONE-Mikroskopie in die Liste der „sieben Technologien, die man 2024 im Auge behalten sollte“ aufgenommen.
Zum ONE-Mikroskopie-Verfahren
Die Auflösung herkömmlicher Lichtmikroskope ist durch die Gesetze der Optik begrenzt: Objekte, die kleiner als 200 Nanometer sind wie zum Beispiel Antikörper mit einer Größe von zirka 15 Nanometern, erscheinen unscharf und können, wenn sie weniger als 200 Nanometer voneinander entfernt liegen, nicht voneinander getrennt sichtbar gemacht werden. Die superauflösende Mikroskopie umgeht diese Beugungsgrenze mit optischen Tricks, so dass Auflösungen von bis zu zehn Nanometer und weniger erreicht werden. Dies erfordert aber sehr teure Mikroskope. Die ONE-Mikroskopie setzt auf eine Vergrößerung des Probenvolumens, um diese Beugungsgrenze zu umgehen. Dabei werden Zellen und die darin enthaltenen Strukturen zunächst chemisch auf ein wasserabsorbierendes Gel gebunden, wie man es in Babywindeln findet. Durch die Aufnahme von Wasser dehnt sich das Gel gemeinsam mit der Probe aus, wodurch sich die einzelnen Moleküle voneinander wegbewegen. Die zusätzliche Einwirkung von Hitze oder Enzymen führt zur Aufspaltung der Proteinmoleküle. Es entstehen einzelne Fragmente, die während der großflächigen Ausdehnung bis um das 15-fache gleichmäßig in unterschiedliche Richtungen bewegt werden, während ihre räumliche Anordnung erhalten bleibt. Eine gezielte Markierung mit fluoreszierenden Molekülen erlaubt es anschließend, die einzelnen Proteinfragmente, die nun in einem Abstand oberhalb der Beugungsgrenze liegen, mit einem herkömmlichen Lichtmikroskop abzubilden. „Wir waren überrascht zu sehen, dass wir so tatsächlich die Y-Form von Antikörpern mit Fluoreszenzmikroskopie sichtbar machen können,“ so Prof. Rizzoli. „Kombiniert mit Künstlicher Intelligenz ist es uns erstmals gelungen, aus zweidimensionalen Fluoreszenzbildern die dreidimensionale Struktur einzelner Proteinmoleküle zu rekonstruieren, ausgehend von der herkömmlichen Lichtmikroskopie.“
Es wurde ein Vergleich der ONE- und der hochauflösenden Kryo-EM-Mikroskopie durchgeführt. Die ermittelte Proteinstruktur des GABAA-Rezeptors, der im Gehirn und im Rückenmark die Aktivität der Nervenzellen steuert, zeigte wie gut die neue Methode funktionierte. „Die ONE-Mikroskopie erlaubt es die gesamte Struktur des GABAA-Rezeptors sichtbar zu machen. Dieser besteht zu 15 bis 20 Prozent aus ungeordneten Schleifen, die mittels der Kryo-EM aus bis zu hunderttausend Bilddaten gemittelt werden müssen. Da diese Schleifen flexible Komponenten sind, das heißt von Rezeptor zu Rezeptor variieren können, lassen sich diese Bilddaten nur unzureichend mitteln. Die Schleifen sind somit nicht erkennbar. Mit der ONE-Mikroskopie liegen bereits weniger als 72 Stunden nach Beginn der Ausdehnung, die ersten Einzelaufnahmen des gesamten Moleküls vor“, sagt Dr. Shaib. „Diese Technik erlaubt es selbst mit einem älteren Lichtmikroskop Auflösungen von mehr als zehn Nanometer zu erreichen. Zum Vergleich, unser Erbgut, die DNA hat einen Durchmesser von zirka 2,5 Nanometer und könnte mit Hilfe dieser Technik dargestellt werden. Jedes Labor, unabhängig von seinen finanziellen Mitteln, kann mit dem ONE-Mikroskopie-Verfahren Bilder in sehr hoher Auflösung erstellen. Dies ist eine Revolution in der Mikroskopie mit langfristigen Auswirkungen, sowohl auf die Wissenschaft als auch auf die Technologie“.
Vielfältiges Anwendungspotential für die Diagnostik
Die ONE-Mikroskopie ermöglicht es Veränderungen der Form von geschädigten oder toxischen Proteinen in menschlichen Proben zu erkennen und bietet damit ein vielfältiges Anwendungspotential. Damit Proteine ihre korrekte Funktion innerhalb der Zelle ausüben können, müssen sie eine dreidimensionale Struktur annehmen. Diese wird durch die Faltung des Proteins erreicht, ein Prozess, bei dem es zu Fehlern kommen kann. Fehlgefaltete Proteine werden entweder abgebaut oder führen zu toxischen Ablagerungen in den Zellen. Hinzu kommt ein Mangel des entsprechenden Proteins und einem damit verbundenen Funktionsverlust in der Zelle und im gesamten Organismus. „Die ONE-Mikroskopie könnte anhand von Blutproben eine visuelle Diagnose von Proteinfehlfaltungserkrankungen wie der Parkinson-Erkrankung ermöglichen,“ sagt Prof. Rizzoli. So gelang es bereits mit Hilfe der ONE-Mikroskopie die Aggregate des Alpha-Synuklein-Proteins in Hirnwasser-Proben von Parkinson-Patient*innen abzubilden und zu klassifizieren. Durch eine Fehlfaltung von Alpha-Synuklein kommt es zur Bildung dieser Aggregate, die sich im Gehirn ablagern und ursächlich für das Absterben von Nervenzellen sind. „Da wir die Form dieser Aggregate leicht erkennen können, besteht die Möglichkeit einer Frühdiagnose dieser neurodegenerativen Erkrankung,“ so Rizzoli. „Dies würde Patient*innen den Zugang zu einer frühzeitigen, wirksamen und personalisierten Behandlung ermöglichen, bevor das Gehirn zu stark geschädigt ist.“
Der Göttinger Exzellenzcluster 2067 Multiscale Bioimaging: Von molekularen Maschinen zu Netzwerken erregbarer Zellen (MBExC) wird seit Januar 2019 im Rahmen der Exzellenzstrategie des Bundes und der Länder gefördert. In einem einzigartigen interdisziplinären Forschungsansatz wird die Entwicklung neuer bildgebender Technologien vorangetrieben, um die krankheitsrelevanten Funktionseinheiten elektrisch aktiver Herz- und Nervenzellen, von der molekularen bis hin zur Organebene abzubilden. Hierfür vereint MBExC zahlreiche universitäre und außeruniversitäre Partner am Göttingen Campus. Das übergeordnete Ziel: den Zusammenhang von Herz- und Hirnerkrankungen zu verstehen, Grundlagen- und klinische Forschung zu verknüpfen, und damit neue Therapie- und Diagnostikansätze mit gesellschaftlicher Tragweite zu entwickeln.
Wissenschaftlicher Ansprechpartner:
Universitätsmedizin Göttingen, Georg-August-Universität
Institut für Neuro- und Sinnesphysiologie
Prof. Dr. Silvio O. Rizzoli
Humboldtallee 23, 37073 Göttingen
silvio.rizzoli@med.uni-goettingen.de
Originalpublikation:
Shaib AH, Chouaib AA, Chowdhury R, Altendorf J, Mihaylov D, Zhang C, Krah D, Imani V, Spencer RKW, Georgiev SV, Mougios N, Monga M, Reshetniak S, Mimoso T, Chen H, Fatehbasharzad P, Crzan D, Saal KA, Alawieh MM, Alawar N, Eilts J, Kang J, Soleimani A, Müller M, Pape C, Alvarez L, Trenkwalder C, Mollenhauer B, Outeiro TF, Köster S, Preobraschenski J, Becherer U, Moser T, Boyden ES, Aricescu AR, Sauer M, Opazo F, Rizzoli SO (2024) One-step nanoscale expansion microscopy reveals protein shapes using conventional microscopes. Nature Biotechnology, 2024. DOI: 10.1038/s41587-024-02431-9.
Weitere Informationen:
https://www.nature.com/articles/s41587-024-02431-9 - Originalpublikation
https://www.rizzoli-lab.de - Institut für Neuro- und Sinnesphysiologie der UMG
https://mbexc.de - Exzellenzcluster „Multiscale Bioimaging: Von molekularen Maschinen zu Netzwerken erregbarer Zellen“ (MBExC)
Die semantisch ähnlichsten Pressemitteilungen im idw