Datenarchivierung mit Molekülen
Synthetische Polymere als Datenspeicher effizienter auslesen
Immer mehr Daten müssen, oft auch langfristig, gespeichert werden. Synthetische Polymere sind eine Alternative zu herkömmlichen Speichermedien, da sie gespeicherte Informationen mit deutlich geringerem Platz- und Energiebedarf aufrechterhalten. Das massenspektrometrische Datenauslesen beschränkt jedoch die Länge und damit die Speicherkapazität der einzelnen Polymerketten. In der Zeitschrift Angewandte Chemie stellt ein Forschungsteam einen neuen Ansatz vor, der diese Limitierung überwindet und einen direkten Zugriff auf interessierende Bits ohne Auslesen der gesamten Kette ermöglicht.
Täglich fallen Daten an, ob im Rahmen geschäftlicher Transaktionen, Prozessüberwachung, Qualitätssicherung oder Rückverfolgbarkeit von Produktions-Chargen. Ihre Archivierung über Jahrzehnte benötigt viel Platz, aber auch Energie. Insbesondere für die langfristige Archivierung großer Datenmengen, auf die nur selten zugegriffen werden muss, sind Makromoleküle mit definierter Sequenz wie DNA und synthetische Polymere eine interessante Alternative.
Gegenüber DNA bieten synthetische Polymere Vorteile: eine einfache Synthese, höhere Speicherdichte und Stabilität unter rauen Bedingungen. Der Nachteil: Die in Polymeren kodierte Information wird durch Massenspektrometrie (MS) bzw. Tandem-Massen-Sequenzierung (MS2) ausgelesen. Hierfür dürfen die Moleküle nicht zu groß werden, was die Speicherkapazität pro Kette stark beschränkt. Außerdem wird die komplette Kette Baustein für Baustein ausgelesen, es kann nicht direkt auf interessierende Bits zugegriffen werden – so als ob man ein Buch, statt auf der relevanten Seite etwas nachzusehen, komplett durchlesen muss. Lange DNA-Ketten können dagegen in Fragmente zufälliger Länge zerlegt, einzeln sequenziert und rechnerisch zur Gesamtsequenz rekonstruiert werden.
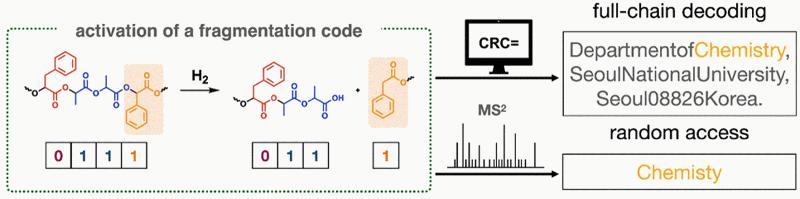
Kyoung Taek Kim und sein Team vom Fachbereich Chemie der Seoul National University (Rep. Korea) entwickelten einen neuen Ansatz, mit dem sehr lange synthetische Polymerketten, deren Molekulargewichte die analytische Grenze der MS bzw. MS2 deutlich überschreiten, effizient ausgelesen werden können. Als Beispiel codierten sie ihre Universitäts-Adresse in einem ASCII-Code und übersetzten diesen – zusammen mit einem Fehler-Detektions-Code (CRC, gängiges Verfahren zur Prüfung der Datenintegrität) – in einen Binärcode, d.h. eine Abfolge von 1 und 0. Die so erzeugte 512-Bit-Information speicherten sie in einer Polymer-Kette aus zwei verschiedenen Monomeren: Milchsäure codiert 1 und Phenyl-Milchsäure 0. An unregelmäßigen Stellen bauten sie zudem Mandelsäure enthaltende Fragmentierungscodes ein. Bei chemischer Aktivierung werden die Ketten dort gespalten, im Beispiel in 18 verschieden große Fragmente, die einzeln durch MS2-Sequenzierung entschlüsselt werden können.
Eine speziell entwickelte Software identifiziert die Fragmente zunächst anhand ihrer Masse sowie ihrer Endgruppen aus den MS-Spektren. Während der MS2 „zerbrechen“ bereits gemessene Molekülionen weiter und die Bruchstücke werden erneut analysiert. Anhand deren Massen-Differenzen lassen sich die Fragmente sequenzieren. Unter Zuhilfenahme der CRC-Fehler-Detektions-Codes rekonstruiert die Software daraus die Sequenz der gesamten Kette. Damit ist die Längen-Limitierung für Polymerketten überwunden.
Außerdem gelang es dem Team, interessierende Bits auch ohne Sequenzierung der gesamten Polymerkette auszulesen (Random Access), z.B. das Wort „Chemistry“ aus dem Code für die Adresse. Unter Berücksichtigung, dass alle Teile der Adresse durch Kommas getrennt und in einer bestimmten Reihenfolge angeordnet sind (Abteilung, Institution, Stadt, Postleitzahl, Land), ließ sich die Stelle eingrenzen, an der die gesuchte Information innerhalb der Kette gespeichert ist, und nur die relevanten Fragmente sequenzieren.
Angewandte Chemie: Presseinfo 16/2024
Autor/-in: Kyoung Taek Kim, Seoul National University (Republic of Korea), http://ktkimgroup.snu.ac.kr/
Angewandte Chemie, Postfach 101161, 69451 Weinheim, Germany.
Die "Angewandte Chemie" ist eine Publikation der GDCh.
Originalpublikation:
https://doi.org/10.1002/ange.202415124
Weitere Informationen:
http://presse.angewandte.de
Ähnliche Pressemitteilungen im idw