Ausstülpungen im Supercomputer berechnet: Wie Zellen ihr internes Kanalsystem verdauen
Im Innern von Zellen gibt es ein weit verzweigtes System von Kanälen, das endoplasmatische Retikulum (ER). Es besteht aus membranumhüllten Röhren, die bei Bedarf – etwa bei Nährstoffmangel – teilweise abgebaut werden. Dazu bilden sich in der Membran Ausstülpungen, die sich dann abschnüren und von der Zelle recycelt werden. Eine Studie der Goethe-Universität Frankfurt hat den Ausstülp-Prozess nun mit Hilfe von Computer-Simulationen untersucht. Demnach spielen dabei bestimmte Struktur-Motive von Proteinen in der ER-Membran eine zentrale Rolle. Die Studie ist im Rahmen der Clusterinitiative „SCALE – Subcellular Architecture of Life“ entstanden.
FRANKFURT. Das Endoplasmatische Retikulum dient unter anderem als Speicher für Kalzium und Kohlenhydrate; außerdem werden in ihm verschiedene Hormone gebildet. Die Zellen passen die Ausdehnung und Vernetzung ihres internen Kanalsystems nach Bedarf an. Eine zentrale Rolle spielt dabei ein Prozess, der sich ER-Phagie nennt (frei übersetzt: „ER-Fressen“). Dabei beult sich ein Teil der Membran um die ER-Röhrchen aus und schnürt sich schließlich als kleines Bläschen ab. Gleichzeitig bildet sich darum eine Art zellinterner Müllsack, das Autophagosom. Dieser verschmilzt dann mit einem anderen Behälter, der hoch reaktive Enzyme enthält. Die Enzyme „schreddern“ den Inhalt des Müllsacks und recyceln ihn.
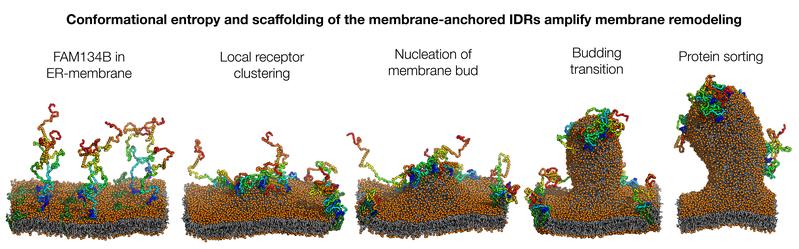
„Seit einigen Jahren ist bekannt, dass bei diesem Prozess bestimmte Proteine eine zentrale Rolle spielen, die ER-Phagie-Rezeptoren“, erklärt Dr. Ramachandra Bhaskara vom Institut für Biochemie II der Goethe-Universität. Diese befinden sich in der Membran um die ER-Röhrchen. Sie bestehen aus einem Anker, mit dem sie in die Membran eintauchen. An diesem Anker sind zwei lange Proteinfäden befestigt, die wie bewegliche Tentakeln von der Membranoberfläche nach außen weisen. „Mittels komplexer Simulationen in Supercomputern konnten wir kürzlich zusammen mit anderen Arbeitsgruppen zeigen, dass der Anker die Membran dazu bringt, sich zu wölben“, sagt Bhaskara. „Unter bestimmten Umständen kann daraus dann eine Ausstülpung entstehen. In der aktuellen Studie haben wir nun nachgewiesen, dass die fädigen Strukturen die Entstehung einer solchen Ausbeulung wahrscheinlicher machen und deutlich beschleunigen.“
Proteine bilden ungeordnete „Tentakeln“ aus Aminosäuren
Die meisten Proteine nehmen nach ihrer Herstellung eine definierte dreidimensionale Gestalt an: Teile von ihnen bilden in sich verdrehte, spiralförmige Strukturen, andere falten sich wie der Balg eines Akkordeons hin und her. Sie erhalten dadurch eine kompakte, relativ starre Form. Das trifft auch für die Anker-Region der ER-Phagie-Rezeptoren zu. Die Tentakeln dagegen bestehen aus langen Ketten von Aminosäuren, die weitgehend regellos hin- und herschlackern. Sie werden daher auch als „intrinsisch ungeordnete (Protein-)Regionen“ oder IDRs (so das englische Kürzel) bezeichnet. Für diese raumgreifenden Bewegungen benötigen sie Platz. Und den verschaffen sie sich, indem sie die Membran, in der sie verankert sind, aufwölben. „Hinzu kommt aber noch ein weiterer Effekt“, betont Dr. Sergio Alejandro Poveda Cuevas, Erstautor der Studie: „Die IDRs enthalten kurze Sequenzen, die sich unter bestimmten Bedingungen sehr wohl falten können. Wir konnten zeigen, dass sie das während der Bildung der Ausbeulungen tun. Sie schmiegen sich dann wie ein Gerüst an die Membran und verstärken ihre Wölbung dadurch noch.“
Die Abschnürung wird also durch verschiedene fein orchestrierte Prozesse eingeleitet, wie die Simulation belegt: Zunächst nähern sich die Anker-Regionen verschiedener ER-Phagie-Rezeptoren einander an. Durch diese Clusterbildung vergrößert sich die von ihnen hervorgerufene Wölbung der Membran. Anfangs sind die IDR-Tentakeln noch ausgefahren. Sie nehmen Kontakt zur Autophagie-Maschinerie auf und dirigieren sie in die Nähe der Membran. Nun kondensieren die IDRs zu kompakteren Strukturen. Dadurch verstärkt sich die Ausbeulung weiter, bis die Membran abgeschnürt und das Bläschen im Autophagosom („Müllsack“) verpackt wird.
Erkenntnisse könnten auch für die Behandlung bestimmter Erkrankungen wichtig sein
„Unsere Studie liefert einen detaillierten Einblick in diesen wichtigen zellulären Prozess“, erklärt Bhaskara. „Sie zeigt zudem, dass die Rezeptor-IDRs für einen reibungslosen Ablauf eine ganz wesentliche Rolle spielen.“ Interessant sind diese Ergebnisse auch deshalb, weil manche angeborenen Erkrankungen der Nervenzellen mit einer gestörten ER-Phagie in Verbindung gebracht werden. Wenn man den Ablauf des Membran-Abbaus besser versteht, erwächst daraus vielleicht irgendwann einmal die Möglichkeit, ihn gezielt zu beeinflussen.
Die Studie wurde durch die Deutsche Forschungsgemeinschaft DFG im Rahmen des Sonderforschungsbereichs 1177 sowie durch das ENABLE-Clusterprojekt des Hessischen Ministeriums für Wissenschaft und Forschung, Kunst und Kultur gefördert.
Video- und Bildmaterial zum Download:
Bilderfolge: https://www.uni-frankfurt.de/160576923
Videoclip (19 MB): https://www.uni-frankfurt.de/160576969
Videobeschreibung/Bildtext: Der ER-Phagie-Rezeptor FAM134B ist anfangs über die Membranoberfläche verteilt (links). Seine IDRs bewegen sich wie Tentakeln weitgehend regellos hin und her. Die Bildung lokaler Cluster leitet die Wölbung der Membran ein (2.v.l.), die sich dann durch die Kondensation der Tentakel zu kompakteren Strukturen (2.v.r.) noch verstärkt. Die Abschnürung des Membranbläschens (nicht gezeigt) wird durch eine gezielte Umverteilung der Proteine („sorting“, rechts) eingeleitet. Bild: AG Bhaskara, Goethe-Universität Frankfurt
Wissenschaftlicher Ansprechpartner:
Dr. Ramachandra M. Bhaskara
Gruppenleiter „Computational Cell Biology“
Institut für Biochemie II
Goethe-Universität Frankfurt
Telefon: +49 (0) 69 6303-2508
bhaskara@med.uni-frankfurt.de
Homepage: https://biochem2.com/research-group/computational-biology
Originalpublikation:
Sergio Alejandro Poveda-Cuevas, Kateryna Lohachova, Borna Markusic, Ivan Dikic, Gerhard Hummer, Ramachandra M. Bhaskara: Intrinsically disordered region amplifies membrane remodeling to augment selective ER-phagy. PNAS (2024) https://doi.org/10.1073/pnas.2408071121
Ähnliche Pressemitteilungen im idw