Familie der „Makler“ hilft beim Aufräumen in der Zelle
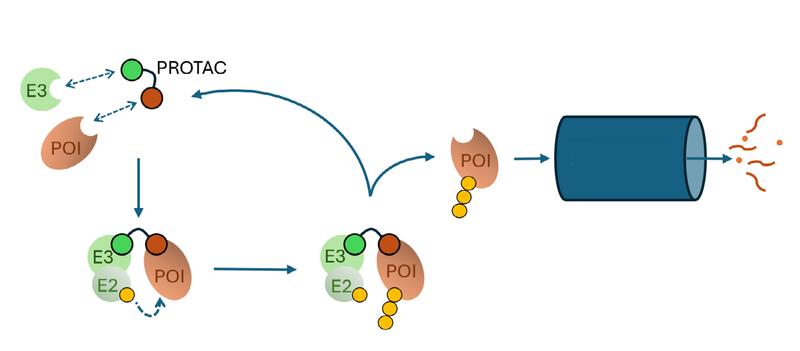
Zellen bilden ständig neue Proteine und bauen nicht mehr benötigte ab. Für den Abbau nutzen die Zellen überwiegend das Ubiquitin-Proteasom-System (UPS), wo Proteine mit Ubiquitin markiert und zu einem „Schredder“ weitergeleitet werden, dem Proteasom. Zentral ist dabei das Enzym E3-Ligase, das als „Makler“ fungiert und die Markierung der abzubauenden Proteine vermittelt. Ein Forschungsteam der Goethe-Universität Frankfurt hat nun erstmals einen systematischen Katalog aller menschlichen E3-Ligasen und ihrer Beziehungen zueinander erstellt und 40 von ihnen als potenzielle Akteure in neu entstehenden Wirkstoffklassen wie den PROTACs identifiziert, die etwa in der Krebstherapie eingesetzt werden.
FRANKFURT. Ordnung zu halten ist eine große logistische Herausforderung für die Zelle: Eine einzelne Säugetierzelle enthält Milliarden von Proteinmolekülen, die präzise gebildet, genutzt und wieder abgebaut werden müssen. Zum Abbau über das Ubiquitin-Proteasom-System (UPS) werden bestimmte Proteine mit Ketten mehrerer Ubiquitin-Moleküle versehen und anschließend von einem Proteasom geschreddert. Der entscheidende Schritt in diesem Prozess ist die Auswahl des spezifischen Zielproteins: E3-Ligasen sind Enzyme, die als molekulare „Makler” an das Zielprotein binden und dann die Übertragung von Ubiquitin von einem E2-Enzym auf das Zielprotein vermitteln.
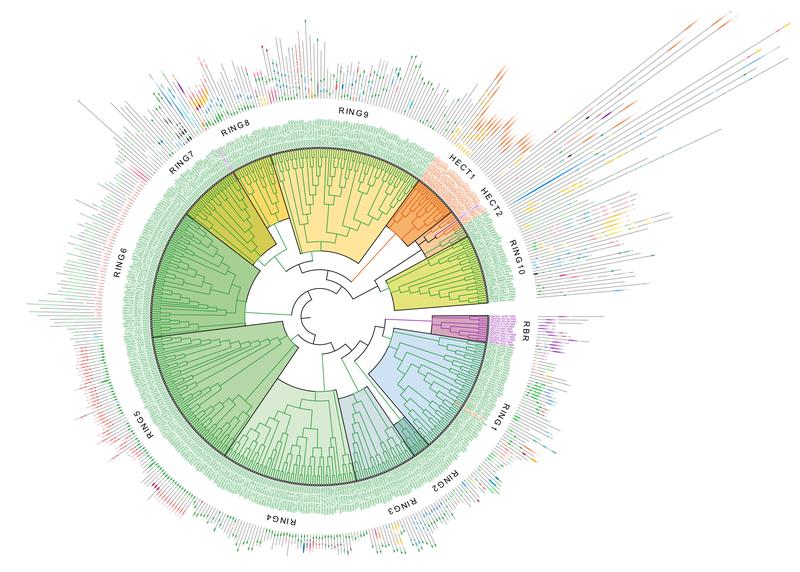
Da jede E3-Ligase nur eine begrenzte Anzahl von Zielproteinen erkennt, verfügen Zellen über ein großes und vielfältiges Repertoire an E3-Ligasen. Ein Forschungsteam der Goethe-Universität Frankfurt unter der Leitung von Dr. Ramachandra M. Bhaskara vom Institut für Biochemie II hat nun alle Mitglieder dieser „Makler-Familie” in einem Katalog zusammengestellt und erstmals aufgezeigt, wie menschliche E3-Ligasen miteinander in Beziehung stehen und was dies für ihre Funktion, die Erkennung der Zielproteine und die Arzneimittelentwicklung bedeutet.
Eine datengestützte Karte des „E3-Ligoms”
Zur Beschreibung dieser Makler-Sippschaft – des sogenannten „E3-Ligoms“ – verglichen die Forschenden im Computer und mit KI-Unterstützung die E3-Ligase-Merkmale miteinander. Anschließend bestätigten sie die Ergebnisse in Zellkulturexperimenten und konnten so Schlussfolgerungen zur Funktion der E3-Ligasen ziehen. Über diese Funktionen definierten sie 13 Hauptfamilien sowie mehrere Unterfamilien, die die Gemeinsamkeiten zwischen den E3-Ligase-Mitgliedern besser wiedergeben als alleinige Aminosäuresequenz- oder Strukturähnlichkeiten. Bhaskara erklärt: „Unser datengestützter Ansatz auf Basis von maschinellem Lernen zeigt Funktionen, die spezifisch für E3-Ligase-Familien sind. So sind die Mitglieder einer Familie beispielsweise wichtig für DNA-Reparaturprogramme und die Vermeidung von ungewolltem Zelltod, während andere an der antiviralen Abwehr beteiligt sind.“
Über ihre Rolle beim Proteinabbau hinaus sind E3-Ligasen auch an Signalübertragungsprozessen beteiligt. Hierzu wird Ubiquitin eingesetzt, das nicht für den Proteinabbau genutzt wird. Dies zeigt, dass E3-Ligasen für zahlreiche zelluläre Signalwege und Krankheitsmechanismen bedeutsam sind.
Bedeutung für Therapeutika der nächsten Generation
Die neue E3-Ligase-Karte ist besonders für neuartige Wirkstoffe wie PROTACs relevant, die den gezielten Proteinabbau ermöglichen. PROTACs (Proteolysis Targeting Chimeras) sind bifunktionelle Moleküle, die eine E3-Ligase in die Nähe eines krankheitsrelevanten Proteins bringen und so dafür sorgen, dass das Protein mit Ubiquitin markiert und durch ein Proteasom geschreddert wird. Obwohl sich das PROTAC-Forschungsgebiet rasch weiterentwickelt hat, stützen sich die meisten bestehenden PROTACs nur auf eine kleine Anzahl gut charakterisierter E3-Ligasen.
Durch die systematische Analyse des gesamten E3-Ligoms identifizierte das Frankfurter Forschungsteam 40 weitere E3-Ligasen, die sich potenziell für die Entwicklung neuer PROTACs eignen. Besonders wichtig: Die E3-Ligase-Karte ermöglicht es, bereits etablierte Bindungspartner von E3-Ligasen für verwandte E3-Ligasen zu verwenden oder anzupassen und Designprinzipien zu übertragen. Auf diese Weise könnte die Entwicklung neuer PROTACs für weitere Körpergewebe, Zellkonstellationen und Krankheiten beschleunigt werden.
Offene Ressource für die Forschungsgemeinschaft
Da weltweit zahlreiche Forschungsgruppen an Ansätzen zum zielgerichteten Proteinabbau arbeiten, hat das Team der Goethe-Universität das vollständige E3-Ligom über eine spezielle Datenbank öffentlich zugänglich gemacht, sodass andere Forschende auf der Klassifizierung und den funktionalen Erkenntnissen aufbauen können.
Wissenschaftlicher Ansprechpartner:
Dr. Ramachandra M. Bhaskara
Gruppenleiter Computational Cell Biology
Institut für Biochemie II
Goethe-Universität Frankfurt
Tel. +49 (0)69 7984-2526
Bhaskara@med.uni-frankfurt.de
https://biochem2.com/people/bhaskara-ramachandra-m
Originalpublikation:
Arghya Dutta, Alberto Cristiani, Siddhanta V. Nikte, Jonathan Eisert, Yves Matthess, Borna Markusic, Cosmin Tudose, Chiara Becht, Varun Jayeshkumar Shah, Thorsten Mosler, Koraljka Husnjak, Ivan Dikic, Manuel Kaulich, Ramachandra M. Bhaskara: Multi-scale classification decodes the complexity of the human E3 ligome. Nature Communications (2025) https://doi.org/10.1038/s41467-025-67450-9
Ähnliche Pressemitteilungen im idw